
Che cos’è il biosensore?
Il biosensore è un dispositivo analitico in grado di identificare le biomolecole, a differenza dei normali sensori, costituito da un elemento sensibile biologicamente attivo e da una parte elettronica, l’elemento biologico interagisce con il substrato da analizzare e un sistema di trasduzione converte la risposta biochimica in un segnale elettrico.
In particolare, convertendo la reazione tra un elemento biologico (elemento biosensibile) come un enzima e un analita in un segnale elettrico, la presenza o l’assenza di una molecola specifica come l’emoglobina o un antigene specifico nel campione al momento dell’esame o Ha la funzione di rilevare e misurare la concentrazione.
Possibilità future nei casi d’uso in campo medico e alimentare
Il ruolo di un “sensore” generale è quantificare valori come calore, luce, radiazione, elettricità, ioni, forza e vibrazione e mostrarli in un formato di facile comprensione.
I sensori vengono utilizzati ovunque nella nostra vita, anche se potremmo non esserne consapevoli. I sensori sono utilizzati nelle porte automatiche, nei forni a microonde e nei frigoriferi.
Con lo sviluppo della biotecnologia, i “biosensori” sono stati utilizzati nei test diagnostici clinici, nell’industria alimentare e nel campo ambientale. Come si è sviluppato il biosensore, che è un analizzatore in grado di identificare le biomolecole, e che tipo di potenziale può essere utilizzato?
Il ruolo di un “sensore” generale è quantificare valori come calore, luce, radiazione, elettricità, ioni, forza e vibrazione e mostrarli in un formato di facile comprensione.
I sensori vengono utilizzati ovunque nella nostra vita, anche se potremmo non esserne consapevoli. I sensori sono utilizzati nelle porte automatiche, nei forni a microonde e nei frigoriferi.
Biosensori per il rilevamento dei virus
Yeşeren Saylan, Özgecan Erdem, Serhat Ünal, Adil Denizli
Abstract
Le malattie infettive rappresentano ancora una minaccia onnipresente per la salute pubblica e globale, soprattutto in molti paesi e nelle aree rurali delle città. Le ragioni alla base di tali gravi malattie possono essere riassunte nella scarsità di metodi di analisi appropriati e di successive strategie di trattamento a causa del limitato accesso a strutture sanitarie centralizzate e attrezzate per la diagnosi. I biosensori hanno un grande impatto per trasformare i nostri attuali metodi analitici in strategie diagnostiche ristrutturando il loro modulo di rilevamento per il rilevamento di biomolecole, in particolare oggetti di dimensioni nanometriche come biomarcatori proteici e virus. Indubbiamente, le attuali piattaforme di rilevamento richiedono aggiornamenti continui per affrontare le crescenti sfide nella diagnosi dei virus poiché i virus cambiano rapidamente e si diffondono ampiamente da persona a persona, indicando l’urgenza di una diagnosi precoce. Alcune delle sfide possono essere classificate in barriere biologiche (specificità, basso numero di bersagli e matrici biologiche) e limitazioni tecnologiche (limite di rilevazione, intervallo dinamico lineare, stabilità e affidabilità), nonché aspetti economici che limitano la loro implementazione in risorse -Impostazioni scarse. In questa recensione, sono stati spiegati in modo completo il principio e i tipi di biosensori e le loro applicazioni nella diagnosi di malattie infettive distinte. L’implementazione degli attuali biosensori in ambienti con scarse risorse viene ulteriormente discussa per il rilevamento dei virus elaborando i pro e i contro dei metodi esistenti come conclusione e prospettiva futura.
Parole chiave: biosensore, applicazioni mediche, rilevamento di virus
- Introduzione
La contaminazione delle fonti da virus è una delle principali cause di malattie che provocano centinaia di migliaia di morti ogni anno. Questi problemi medici non sono stati ancora risolti, come dimostrano milioni di persone che soffrono di diverse malattie [1]. Al giorno d’oggi, c’è un notevole aumento dell’incidenza di malattie infettive che hanno un effetto importante su tutte le specie vive (animali, umani e piante) [2]. Soprattutto in molti paesi e segmenti poveri della società moderna, diverse malattie contagiose come la tubercolosi, la malaria e il virus dell’immunodeficienza umana stanno colpendo molte persone e continuano anche a creare significativi problemi di salute [3]. I virus sono parassiti intracellulari obbligati e hanno bisogno della cellula ospite per propagarsi e stabilire la replicazione del materiale genetico. I loro complicati meccanismi di protezione possono cambiare molto rapidamente. In risposta a questa situazione, i virus si adattano abbattendo e manipolando la reazione immunitaria dell’ospite. Ciò ha portato all’emergere di virus che si adattano a manipolare e sovvertire le risposte immunitarie dell’ospite. Inoltre, le infezioni virali causano morbilità e mortalità in tutto il mondo. In particolare, alcuni focolai hanno attirato l’attenzione negli ultimi anni; sottotipo di influenza A H1N1 nel 2009 e epidemia di virus Ebola nel 2014 [4].
La determinazione precoce di agenti patogeni come batteri e virus è cruciale per scopi clinici point-of-care [5]. La reazione a catena della polimerasi [6], il test di immunoassorbimento enzimatico [7], la reazione a catena della polimerasi a trascrizione inversa [8] e diverse tecnologie di biosensori vengono utilizzate per rilevare o rimuovere i virus [9,10,11,12,13]. Sul mercato si possono trovare strumenti di rilevamento dei virus basati su antigene-anticorpo o recettore-ligando. Indipendentemente dalle loro numerose circostanze favorevoli, le biomolecole possiedono difetti essenziali in termini di utilizzazione e stabilità [14]. A causa della necessità di una diagnosi rapida e degli sviluppi nella tecnologia dei biosensori più selettiva, stabile ed economica, sono stati studiati nuovi elementi di riconoscimento per migliorare il riconoscimento nel biosensore [15]. I biosensori sono dispositivi analitici costituiti da un analita, un biorecettore, un trasduttore e un segnale misurabile [16]. L’analita viene catturato e le risposte biologiche vengono convertite in segnali. Per quanto riguarda i principi di trasduzione, i biosensori possono essere classificati in tre classi principali come ottici [17,18,19], elettrochimici [20,21,22] e piezoelettrici [23,24,25,26]. Negli ultimi due decenni, i biosensori si sono rivelati importanti per rilevare analiti distintivi, ad esempio esplosivi [27,28], proteine [29,30], acidi nucleici [31,32], biomarcatori del cancro [33,34] , batteri [35,36], virus [37,38] e tossine [39,40] nella lavorazione degli alimenti [41], nel monitoraggio ambientale [42], nella diagnostica clinica [43,44] e nella lotta al bioterrorismo [45] .
In questa recensione, vengono prima spiegati il principio e i tipi di biosensori, quindi vengono ampiamente discussi i nuovi tipi e le nuove tecnologie di biosensori in base alle ultime scoperte della ricerca per la valutazione rapida delle applicazioni mediche alla diagnosi delle malattie infettive. Infine, vengono citate le conclusioni e le prospettive future per confrontare i biosensori commerciali e riassumere la revisione.
- Principio del biosensore
Un biosensore è un dispositivo analitico con tre moduli principali: (i) un biorecettore sensibile; (ii) un trasduttore; e (iii) un rivelatore con un’uscita digitale. Principalmente, l’analita bersaglio interagisce con il biorecettore [46] e la parte componente di rilevamento riconosce specificamente l’analita attraverso una reazione, un adsorbimento specifico o un altro processo come l’interazione fisica/chimica. Quindi, il trasduttore traduce i cambiamenti molecolari in un segnale quantificabile misurato dal modulo del rivelatore digitale [47]. L’argomento dei principi di trasduzione può essere separato come elettrochimico, piezoelettrico, ottico, termico, micromeccanico e magnetico. I biosensori forniscono molteplici funzionalità, tra cui prestazioni eccezionali, funzionamento intuitivo, risposta rapida, elevata sensibilità e specificità, portabilità, dimensioni relativamente compatte e analisi in tempo reale [48]. Oggigiorno, i ricercatori intendono migliorare la sensibilità e la specificità delle tecniche concentrandosi sullo sviluppo dei biosensori e sulla qualità della fabbricazione, ampliando l’affinità tra la creazione di chimiche superficiali innovative e l’utilizzo di nanomateriali come nanofilm [49], nanoparticelle [50] o punti quantici [ 51] per l’amplificazione del segnale.
2.1. Tipi di biosensori
2.1.1. Biosensori elettrochimici
I biosensori elettrochimici sono stati utilizzati per lungo tempo per raggiungere un’ampia gamma di applicazioni in vari settori. Questi biosensori rappresentano una tipica piattaforma per la costruzione di biosensori, che includono semiconduttori ed elettrodi serigrafati [52]. In breve, questi biosensori monitorano eventuali alterazioni nelle proprietà dielettriche, nella dimensione, nella forma e nella distribuzione di carica mentre il complesso antigene-anticorpo si forma sulla superficie dell’elettrodo. Possono essere classificati in quattro gruppi principali tra cui trasduttori potenziometrici, amperometrici, voltammetrici ciclici e impedimetrici [53]. Questi biosensori sono stati impiegati per rilevare una varietà di bersagli biologici, tra cui proteine, biomarcatori del cancro, acido nucleico e così via [54,55,56].
2.1.2. Biosensori piezoelettrici
Uno dei biosensori piezoelettrici più comuni è il biosensore a microbilancia a cristallo di quarzo, che misura qualsiasi cambiamento di massa e viscoelasticità dei materiali registrando la frequenza e il cambiamento di smorzamento di un risonatore a cristallo di quarzo [57]. A causa dell’elevata sensibilità alle condizioni ambientali, il meccanismo di rilevamento richiede in modo significativo apparecchiature di isolamento che riducano al minimo eventuali fattori di ostacolo come le vibrazioni. Questi biosensori sono stati utilizzati in un’ampia varietà di applicazioni per rilevare bersagli inclusi ormoni, batteri, cellule e così via [58,59,60].
2.1.3. Biosensori ottici
I biosensori ottici si concentrano sulla misurazione di un cambiamento nelle caratteristiche ottiche della superficie del trasduttore quando l’analita e l’elemento di riconoscimento formano un complesso. Questi biosensori possono essere suddivisi in due gruppi. Ad esempio, la generazione di un segnale dipende dalla formazione di un complesso sulla superficie del trasduttore nel biosensore ottico diretto. I biosensori ottici indiretti sono principalmente progettati con varie etichette come fluorofori o cromofori per rilevare gli eventi di legame e amplificare il segnale. Sebbene i metodi di biorilevamento indiretto possano produrre livelli di segnale più elevati, risentono del legame non specifico e dell’elevato costo del reagente della fase di etichettatura [61]. Nella letteratura e nel mercato sono presenti molteplici biosensori ottici, inclusi biosensori ottici in fibra a base di optrodi, biosensori ottici in fibra ad onda evanescente, fluorescenza risolta nel tempo, biosensore ottico a specchio risonante, biosensori interferometrici e biosensori a risonanza plasmonica di superficie. La loro finestra di rilevamento è così versatile e rilevano più tipi di biomolecole da campioni fisiologici e biologici [62].
2.2. Importanza dei biosensori nella diagnosi medica
L’innovazione dei biosensori si è sviluppata molti anni fa. Diversi ricercatori tra cui biologi, chimici, fisici, medici si sono uniti per utilizzare il biosensore come un’applicazione originale in diversi campi come l’analisi del doping [63], la diagnosi [64], la sicurezza alimentare [65], la medicina di laboratorio [66], e così via. Tra questi, certamente, gli esami clinici sono stati ulteriormente esplorati come una potente area di applicazione. A causa della necessità di analisi rapide e miglioramenti nel rilevamento delle caratteristiche, ovvero stabilità, selettività ed essere redditizi, sono stati studiati nuovi elementi di riconoscimento e disposizioni di essi per migliorare il riconoscimento nei sistemi di biosensori. L’innovazione di nuovi componenti di riconoscimento e l’utilizzo della nanotecnologia hanno contribuito al miglioramento dei biosensori. Le prestazioni analitiche dei biosensori sono aumentate nel rilevamento delle caratteristiche con questa combinazione. Questi punti salienti rendono i biosensori appropriati per la diagnostica point-of-care perché possono ottenere un rilevamento rapido e multi-analita [67]. Esistono diversi tipi di biosensori tra cui ottico [68,69], elettrochimico [70,71], piezoelettrico [72,73], magnetico [74,75], micromeccanico [76,77] e termico [78,79,80 ,81] per la diagnosi medica.
- Ultime applicazioni di biosensori sul rilevamento di virus
3.1. Virus dell’immunodeficienza umana
Il virus dell’immunodeficienza umana (HIV) è un membro di un sottogruppo di retrovirus denominati lentivirus. I lentivirus significano anche un virus lento; e indica il periodo tra l’inizio dell’infezione e l’insorgere dei sintomi. L’HIV infetta le cellule T CD4+ e inizia a replicarsi rapidamente dopo essere entrato nel flusso sanguigno [82]. La fase finale della sindrome da immunodeficienza acquisita da infezione (AIDS) è uno dei problemi di salute pubblica in sospeso. Secondo l’Organizzazione Mondiale della Sanità, finora più di 35 milioni di persone sono state infettate dall’HIV. Nel 2017, 940.000 persone sono morte a livello globale per cause legate all’HIV [83]. C’è un urgente bisogno di una piattaforma di biorilevamento più sensibile e specifica per diagnosticare l’HIV. Esistono due tipi di virus HIV e l’HIV-1 è il tipo più comune che causa la malattia. Ci sono diversi studi di ricerca recenti su questo rilevamento di virus tramite biosensori. Per esempio; Babamiri et al. ha sviluppato un biosensore a elettro-chemiluminescenza basato su imprinting per il rilevamento del gene dell’HIV-1 [84]. Hanno usato l’aptamer dell’HIV come modello e l’o-fenilendiammina come monomero funzionale (Figura 1a). Dopo gli esperimenti, hanno osservato che la risposta aumentava significativamente dopo la reazione di ibridazione. Hanno ottenuto un rilevamento del gene HIV molto sensibile (0,3 fM) in un intervallo da 3,0 fM a 0,3 nM. Il biosensore preparato ha mostrato una buona specificità per il rilevamento dell’HIV rispetto alle sequenze non complementari. Hanno anche testato campioni di siero e ottenuto recuperi elevati nell’intervallo del 95-101,2%.
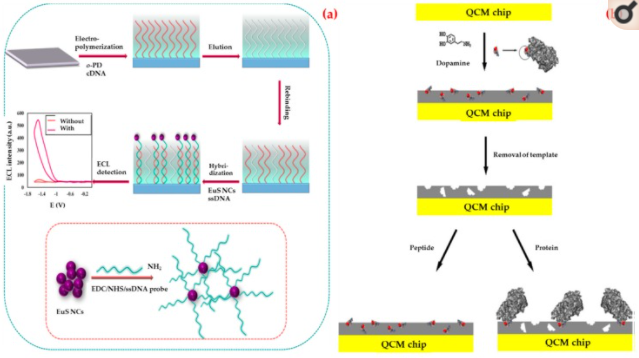
Rappresentazioni schematiche dell’elettro-chemiluminescenza (a) e della microbilancia dei cristalli di quarzo; (b) biosensori per la rilevazione dell’HIV-1. Ripubblicato con il permesso di [84,85]; autorizzazione trasmessa tramite Copyright Clearance Center, Inc.%.
La glicoproteina41 (Gp41) è la proteina transmembrana dell’HIV-1 e ha un ruolo significativo nella fusione della membrana tra cellule infette e virus. L’entità della progressione dell’AIDS e l’efficacia dell’intervento terapeutico possono essere monitorate con Gp41. Lu et al. ha sviluppato un biosensore con lo scopo di rilevare la Gp41 correlata all’HIV-1 [85]. Hanno modificato una superficie del biosensore della microbilancia a cristalli di quarzo con un peptide sintetico che è analogo a 579-613 residui di Gp41 mediante il metodo di imprinting dell’epitopo (Figura 1b). Secondo i risultati, hanno mostrato che un film impresso ha una grande affinità con il peptide bersaglio e può legare selettivamente la proteina Gp41. Hanno anche scoperto che il limite di rilevamento è di 2 ng/mL.
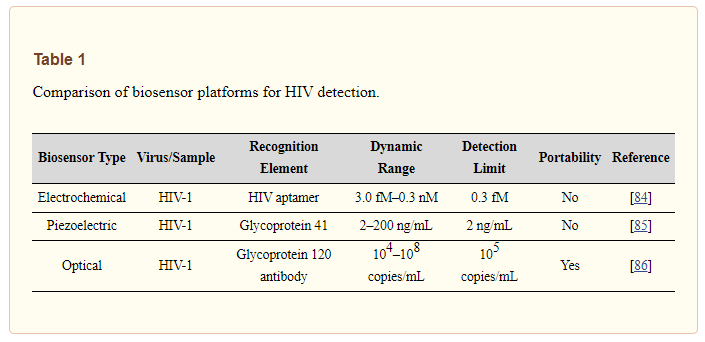
È stato dimostrato che una piattaforma di rilevamento ottico rileva l’HIV-1 da campioni biologici da Shafiee et al. [86]. In questo studio, hanno osservato che quando il virus intatto veniva adsorbito dalla superficie, questa situazione induceva uno spostamento nel valore della lunghezza d’onda del picco di risonanza. Questo valore può essere rilevato con una risoluzione della lunghezza d’onda di 10 pm. Con questo biosensore è possibile rilevare sia gli strati biomolecolari che anche basse concentrazioni di virus. Hanno anche esaminato il rilevamento dell’HIV-1 in campioni di siero e soluzione salina tamponata con fosfato con cariche virali comprese tra 104 e 108 copie/mL. La tabella 1 fornisce un confronto delle piattaforme di biosensori per il rilevamento del virus dell’immunodeficienza umana.
3.2. Epatite
È noto almeno dagli anni ’40 che campioni di sangue e plasma possono contenere un virus che causa epatite transitoria e cronica, nota anche come epatite post-trasfusione [87]. L’epatite B è una delle principali infezioni dell’umanità, che si stima causi circa 800.000 decessi all’anno, per la maggior parte a causa della crescita maligna del fegato e della cirrosi. Quasi il 15-40% dei pazienti infetti svilupperà insufficienza epatica, cirrosi epatica o carcinoma epatocellulare e il 15-25% alla fine morirà [88].
Hassen et al. hanno riportato un metodo basato sull’ibridazione del DNA per rilevare il virus dell’epatite B utilizzando la spettroscopia di impedenza elettrochimica non faradica [89]. Hanno prima modificato le sonde del DNA con biotina su nanoparticelle magnetiche a base di streptavidina e poi hanno immobilizzato le nanoparticelle sull’elettrodo d’oro nudo usando un magnete. Dopo gli esperimenti di caratterizzazione, hanno dimostrato che l’immobilizzazione delle sonde del DNA e l’ibridazione con diverse concentrazioni di DNA complementare hanno avuto successo. Inoltre, hanno dimostrato che la spettroscopia di impedenza non faradica può essere rilevata 50 picomoli di HBV DNA su un campione di 20 μL e la saturazione è stata raggiunta 12,65 nmole/mL per la stessa quantità di sonde di DNA immobilizzate.
Inoltre, Tam et al. ha studiato la rilevazione della capacità dell’anticorpo dell’antigene di superficie dell’epatite B utilizzando un biosensore di risonanza plasmonica di superficie [90]. Hanno ottenuto una prestazione lineare in un intervallo di 0,00098-0,25 mg/L e un limite di valore di rilevamento sette volte più elevato, ovvero un aumento di due volte del coefficiente di varianza dei risultati replicati, rispetto all’immunosorbente legato all’enzima. saggio. Inoltre, hanno riferito che la valutazione del dosaggio per la specificità non presentava reattività crociata con altri anticorpi testati.
Uzu et al. ha anche rilevato un anticorpo di superficie dell’epatite B che impiega un biosensore di risonanza plasmonica di superficie per la diagnosi di epatite nel siero umano [91]. Hanno eseguito studi cinetici utilizzando campioni di siero umano positivi agli anticorpi di superficie dell’epatite B (Figura 2a). Secondo i calcoli matematici, hanno mostrato che questo biosensore aveva una certa omogeneità superficiale, obbediva al modello isotermico di adsorbimento di Langmuir e aveva un valore limite di rilevamento basso (208,2 milli-unità internazionale per millilitro, mIU/mL). Infine, hanno eseguito esperimenti di controllo utilizzando campioni di siero non immunizzati (anticorpo di superficie dell’epatite B negativo) ei risultati hanno mostrato che il biosensore non ha dato alcuna risposta evidente al siero negativo.
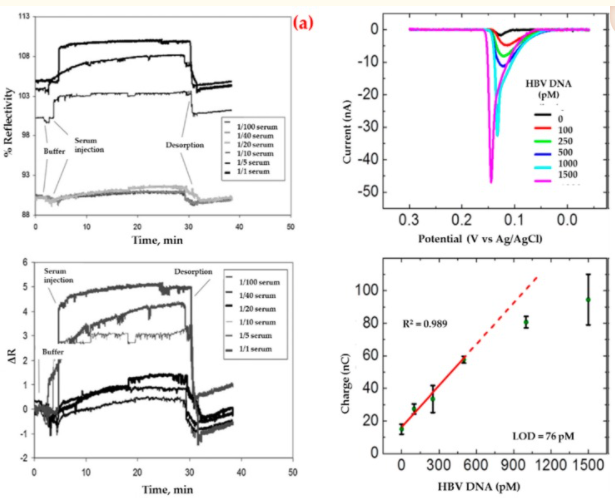
Sensorgrammi per l’interazione tra siero umano positivo agli anticorpi di superficie dell’epatite B e biosensore (a) e voltamogrammi per il test sandwich del DNA del virus dell’epatite B in un biosensore elettrochimico convenzionale (b). Ripubblicato con il permesso di [91,92].
D’altra parte, Li et al. ha sviluppato un biosensore impedimetrico modificato con nanoparticelle d’oro per l’ibridazione selettiva del DNA correlata al virus dell’epatite B [92]. Hanno presentato che il biosensore aveva un alto coefficiente di correlazione in un intervallo di bassa concentrazione (Figura 2b), risposte ripetibili e forniva una superficie adatta per un maggiore legame al DNA. Inoltre, è stata studiata la selettività di questo biosensore in presenza del bersaglio e delle altre sequenze di DNA.
Istek et al. ha preparato un biosensore elettrochimico su carta per rilevare il DNA del virus dell’epatite B [93]. Hanno riferito che questo biosensore aveva quattro attributi necessari. Prima di tutto, hanno combinato il design con la piegatura della carta per l’incubazione dei tempi. Nella seconda parte sono state eseguite due fasi di amplificazione; nanoparticelle d’argento marcate è stato fornito un fattore di amplificazione massimo e microsfere magnetiche sono state catturate le sonde. In terzo luogo, il fatto che nello studio non sia stato utilizzato alcun enzima o anticorpo ha migliorato la stabilità, la velocità e la robustezza del biosensore. Infine, è stato necessario un solo passaggio di incubazione del campione prima di iniziare il rilevamento. Hanno trovato il valore limite di rilevamento a 85 pM.
Zengin et al. ha riportato un biosensore per la sequenza del DNA del rilevamento del virus dell’epatite B che dipendeva da un test sandwich e dallo scattering Raman potenziato in superficie. In primo luogo, hanno preparato un substrato di silicio ibrido sensibile alla temperatura per immobilizzare il filamento di DNA sulla superficie delle nanoparticelle d’oro. Quindi, è stata eseguita la strategia sandwich per il rilevamento del DNA bersaglio con segnali di diffusione Raman ad alta superficie potenziata. Hanno misurato le concentrazioni di DNA del virus dell’epatite B più basse a livelli fM a diverse temperature. Hanno affermato che questa piattaforma altamente sensibile e robusta può essere estesa per rilevare altre biomolecole e specie chimiche senza alcuna etichettatura [94].
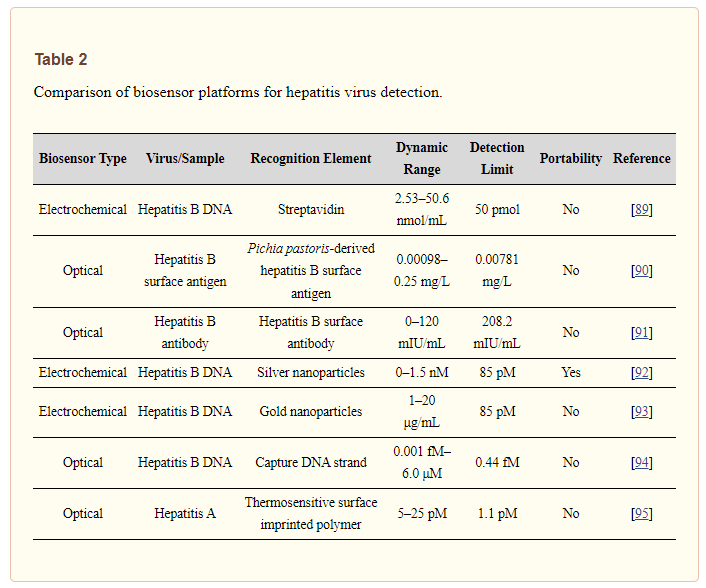
Liu et al. ha sviluppato un biosensore a base di polimeri termosensibili impressi sulla superficie per il monitoraggio del virus dell’epatite A. Hanno regolato le prestazioni di riconoscimento del biosensore mediante il controllo della temperatura per la cattura del virus. Inoltre, hanno calcolato un valore limite di rilevamento molto basso (1,1 pM). Hanno anche utilizzato con successo questo biosensore per rilevare il virus dell’epatite A aggiuntivo da una diluizione di siero umano e hanno ottenuto recuperi elevati nell’intervallo dal 90,8 al 108,3% a tre livelli di virus. Infine, hanno affrontato i problemi dell’elevato adsorbimento non specifico e della lunga durata del processo di rilevamento [95]. Per riassumere e confrontare questi studi, la Tabella 2 fornisce informazioni dettagliate sulle piattaforme di biosensori per il rilevamento del virus dell’epatite.
3.3. Ebola
Le infezioni da Ebola causano gravi malattie negli esseri umani. I pazienti hanno effetti collaterali simil-influenzali generali prima di una rapida progressione della malattia che è caratterizzata da insufficienza multiorgano, emorragia e una sindrome simile allo shock dopo un tempo di incubazione (3-21 giorni) [96]. La più grande epidemia di infezione da virus Ebola ha registrato 15.935 casi segnalati e 5689 decessi nel 2014 [97]. Tali vantaggi innovativi sarebbero ideali per l’identificazione rapida e la determinazione del virus Ebola al punto di cura, in particolare in ambienti con infrastrutture insignificanti, ad esempio, in condizioni di benessere generale o di risposta alle emergenze [98].
Ad esempio, Ilkhani et al. ha fabbricato un biosensore elettrochimico per la diagnostica del DNA del virus Ebola mediante un rilevamento amplificato da un enzima [99]. Come illustrato nella Figura 3a, hanno etichettato l’ibrido biotinilato con un coniugato streptavidina-fosfatasi alcalina. Hanno ottimizzato tutte le fasi dell’esperimento utilizzando la spettroscopia di impedenza elettrochimica e quindi hanno ottenuto un valore limite di rilevamento basso (4,7 nM) utilizzando questo biosensore e la deviazione standard della soluzione del bianco. Hanno infine eseguito la selettività e la riproducibilità del biosensore elettrochimico.
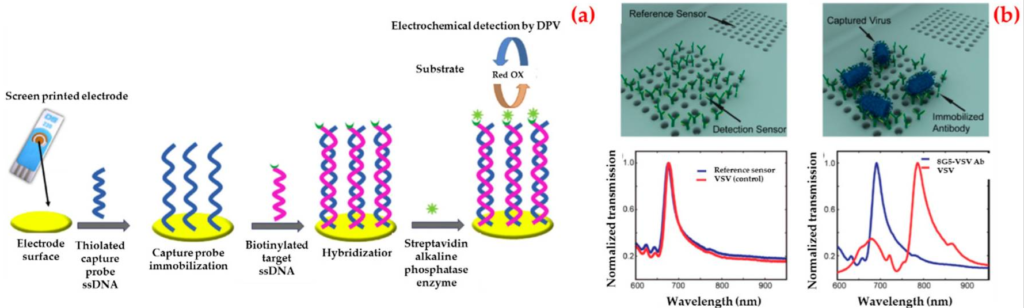
La rappresentazione schematica delle diverse fasi per la fabbricazione del biosensore elettrochimico (a) e i rendering tridimensionali e le misurazioni sperimentali illustrano lo schema di rilevamento utilizzando biosensori nanoplasmonici optofluidici basati su trasmissioni di risonanza dovute allo straordinario effetto di trasmissione della luce (b)
Inoltre, Yanuk et al. ha dimostrato una piattaforma di biosensori optofluidici che ha rilevato direttamente virus interi da mezzi biologici [100]. Il rilevamento può essere effettuato a concentrazioni clinicamente correlate ed è necessaria la preparazione del campione. Il biosensore preparato dipendeva da un impatto sulla trasmissione della luce nei nanofori plasmonici e utilizzava anticorpi specifici del gruppo. Hanno avvolto il rilevamento di piccoli virus a RNA (virus della stomatite vescicolare ed Ebola pseudotipizzata) all’interno di un intervallo dinamico che copre tre ordini di grandezza. Per questi esperimenti, hanno immobilizzato gli anticorpi contro la glicoproteina Ebola sui biosensori e gli spettri di trasmissione sono stati raccolti dopo il processo di lavaggio (Figura 3b).
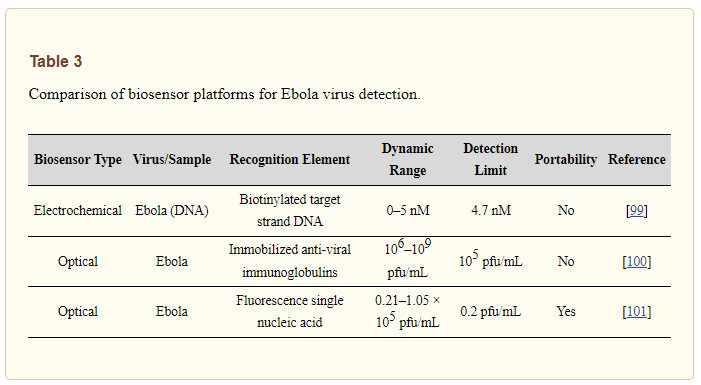
Cai et al. ha anche riportato uno studio sulla rilevazione e quantificazione senza amplificazione del virus Ebola su campioni clinici [101]. Un chip microfluidico è stato utilizzato per la preparazione del campione e la pre-concentrazione del virus. Dietro questo passaggio, il rilevamento della fluorescenza dell’acido nucleico singolo in guide d’onda ottiche a nucleo liquido su un chip di silicio è stato eseguito entro dieci minuti. Hanno anche dimostrato che questo biosensore aveva un’eccezionale specificità e un basso limite di valore di rilevamento (0,2 pfu/mL). C’è un confronto delle piattaforme di biosensori nella Tabella 3 per il rilevamento del virus Ebola.
3.4. Zika
Il virus Zika è un virus trasmesso dalle zanzare identificato per la prima volta in Uganda. Prima del 2007, solo sporadici casi di malattia umana venivano registrati da nazioni in Africa e in Asia. Il primo focolaio documentato di infezione da virus Zika è stato segnalato negli Stati Federati di Micronesia nel 2007 [102]. Le malattie infettive emergenti, tra cui l’epidemia di virus Ebola nell’Africa occidentale in corso, e ora l’epidemia di virus Zika che attraversa l’emisfero occidentale, hanno rinnovato la necessità di creare test diagnostici semplificati da utilizzare in contesti con risorse limitate [103].
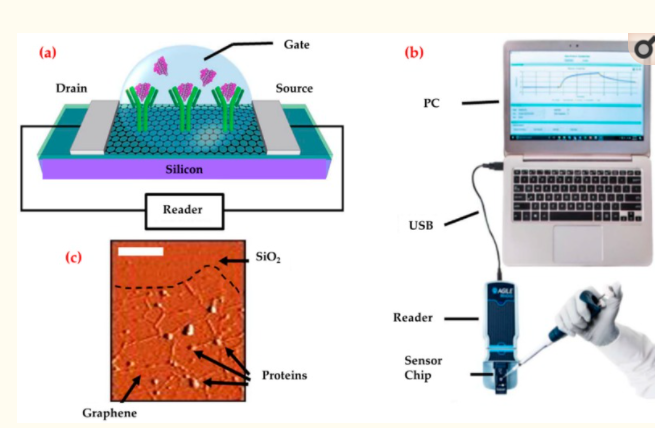
Un biosensore redditizio e portatile abilitato al grafene è stato sviluppato da Afsahi et al. per rilevare il virus Zika con un anticorpo monoclonale immobilizzato altamente specifico [104]. Hanno legato in modo covalente gli anticorpi monoclonali al grafene per il rilevamento degli antigeni virali Zika nativi (Figura 4). Hanno misurato basse concentrazioni di antigeni (450 pM). Hanno anche dimostrato potenziali applicazioni diagnostiche misurando l’antigene Zika in un siero umano ricreato e le prestazioni cliniche del biosensore misurando l’antigene Zika nel siero umano e hanno anche convalidato la selettività con l’encefalite giapponese NS1.
Inoltre, Kaushik et al. ha presentato un biosensore elettrochimico per il rilevamento della proteina del virus Zika [105]. Hanno eseguito la spettroscopia di impedenza elettrochimica per misurare la risposta elettrica del biosensore in funzione delle concentrazioni di proteine e hanno mostrato che questo biosensore ha rilevato selettivamente la proteina del virus Zika in un intervallo di rilevamento da 10 pM a 1 nM e un valore limite di rilevamento di questo biosensore inferiore a 10 pM ad alta sensibilità.
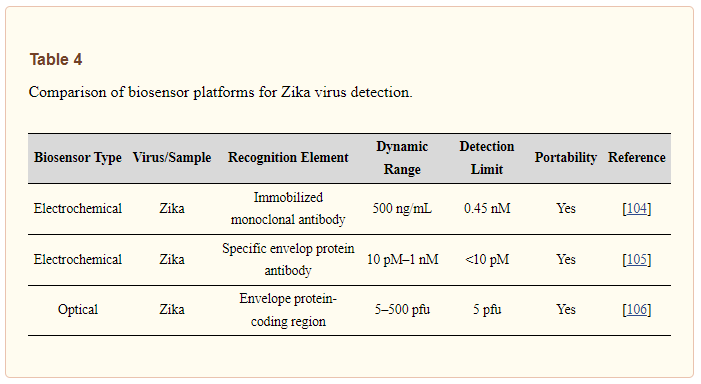
Canzone et al. ha anche riportato un saggio di amplificazione isotermica mediata da loop di trascrizione inversa (RT-LAMP) per la diagnosi del virus Zika come cassetta monouso [106]. Una tazza riscaldata chimicamente è stata utilizzata per il controllo termico della cassetta. Per questo motivo non era necessaria energia elettrica. I ricercatori hanno progettato una serie di nuovi primer RT-LAMP per il virus Zika, la regione codificante le proteine dell’involucro. Hanno dimostrato l’utilità di questo sistema diagnostico rilevando il virus in campioni orali con una sensibilità di 5 pfu in meno di 40 minuti. La tabella 4 confronta le piattaforme di biosensori per il rilevamento del virus Zika.
3.5. Norovirus
Norovirus, è un patogeno enterico umano che causa una malattia significativa in tutta l’assistenza sanitaria. Il norovirus è un importante motivo di morbilità a causa della gastroenterite acuta sia all’interno delle istituzioni sanitarie che nella rete più estesa [107]. La contaminazione da Norovirus avviene generalmente da acqua o cibo contaminati e l’infezione può facilmente diffondersi da persona a persona attraverso le vie fecali o orali [108]. La ragione più importante per epidemie di gastroenterite acuta e malattie sporadiche è stata identificata come Norovirus in tutto il mondo [109]. Numerosi esami si sono concentrati nel fornire tecniche alternative promettenti per rilevare i norovirus in modi più sensibili e accurati, come biosensori elettrochimici basati su aptameri e chip microfluidici.
Esemplare, Ashiba et al. ha studiato un biosensore di risonanza plasmonica di superficie per rilevare particelle simili al virus del norovirus [110]. Hanno progettato questo biosensore che utilizzava un chip dotato di una trincea a forma di V (Figura 5a). Hanno selezionato una lunghezza d’onda di eccitazione di 390 nm per eccitare la risonanza plasmonica superficiale su una pellicola di alluminio del biosensore. Hanno calcolato la concentrazione minima rilevabile come 0,01 ng/mL, che corrisponde a 100 particelle simili a virus incluse nella regione di rilevamento del V-trench.
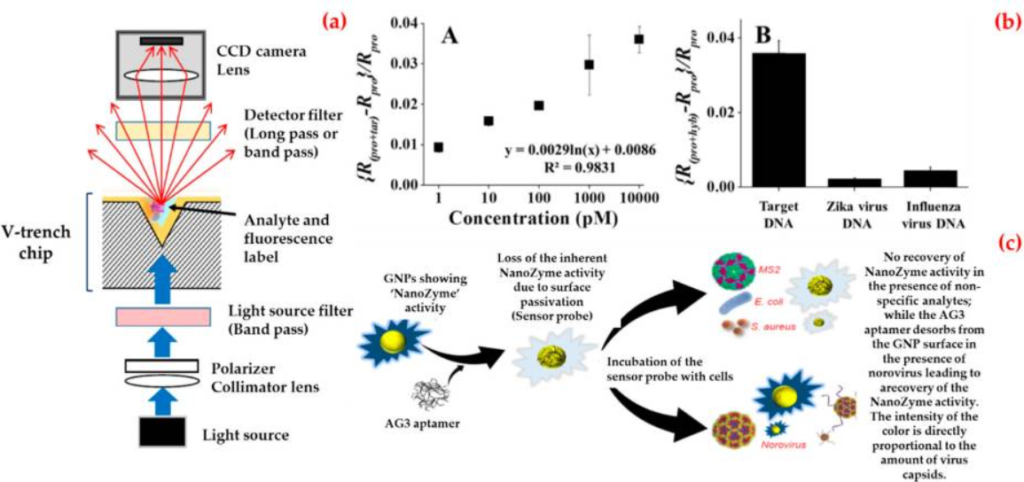
Schema schematico di un biosensore V-trench (a); rilevamento del DNA del norovirus: test di sensibilità e specificità (b); e principio di funzionamento del biosensore per il rilevamento del norovirus (c). Ripubblicato con il permesso di [110,111,112].
Inoltre, Lee et al. sintetizzati nanotubi di carbonio decorati con nanoparticelle binarie e applicati come piattaforma di biorilevamento [111]. Per prima cosa si sono allineati su un elettrodo interdigitato di platino con nanoparticelle d’oro/nanoparticelle magnetiche e nanotubi di carbonio, quindi hanno attaccato un DNA di sonda funzionalizzato con gruppi tiolici alla superficie delle nanoparticelle d’oro per ottenere questa struttura ibrida. Come mostrato nella Figura 5b, hanno monitorato diverse concentrazioni di DNA target (1 pM-10 nM) e hanno calcolato il limite del valore di rilevamento intorno a 8,8 pM. Hanno anche confermato la specificità utilizzando altre sequenze di DNA non corrispondenti.
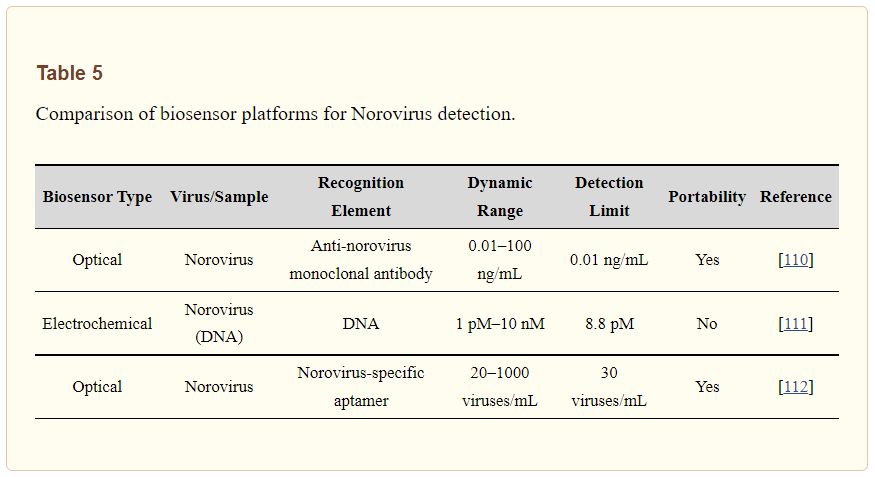
Inoltre, Weerathunge et al. ha proposto una recente piattaforma di biosensori colorimetrici per il rilevamento rapido e ultrasensibile del norovirus murino infettivo [112]. Hanno combinato l’attività catalitica mimica enzimatica delle nanoparticelle d’oro con un’elevata specificità bersaglio di un aptamero di norovirus murino per creare un biosensore e hanno osservato che questo biosensore produceva un colore blu in presenza di norovirus (Figura 5c). Hanno anche calcolato il limite del valore di rilevamento come tre virus per test pari a 30 virus/mL di campione e il limite di rilevamento dimostrato sperimentalmente come 20 virus per test pari a 200 virus/mL. Inoltre, hanno dimostrato la robustezza del biosensore del norovirus testandone le prestazioni in presenza di altri microrganismi nel siero umano e nell’omogenato di crostacei. La tabella 5 fornisce un confronto tra piattaforme di biosensori per il rilevamento di Norovirus.
3.6. Influenza
L’influenza è una malattia infettiva virale considerata come una fonte di numerosi problemi medici e un enorme onere finanziario [113]. Le tecniche regolari sono carenti per il rilevamento sul campo delle infezioni e generalmente sono difficili e richiedono tempo. Di conseguenza, sono immediatamente necessarie ricerche che puntano a migliorare alternative efficaci alle tecniche convenzionali [114].
Ad esempio, Sayhi et al. ha sviluppato un metodo con l’obiettivo di isolare e rilevare il sottotipo H9N2 del virus dell’influenza A [115]. Per prima cosa hanno attaccato un anticorpo proteico anti-matrice 2 a nanoparticelle magnetiche di ferro e le hanno usate per isolare il virus dell’influenza da un fluido allantoico. Successivamente, la fetuina A è stata attaccata a un’etichetta rilevabile elettrochimicamente, le nanoparticelle d’oro, per rilevare il vantaggio dell’attaccamento del virus dall’interazione fetuina-emoagglutinina (Figura 6a). Hanno isolato il complesso e lo hanno trattato con una soluzione acida per raccogliere nanoparticelle d’oro per la deposizione su un elettrodo di carbonio serigrafato. Hanno riferito che questo biosensore consente il rilevamento rapido del virus dell’influenza A/H9N2 a un titolo inferiore a 16 unità di emoagglutinina (HAU).
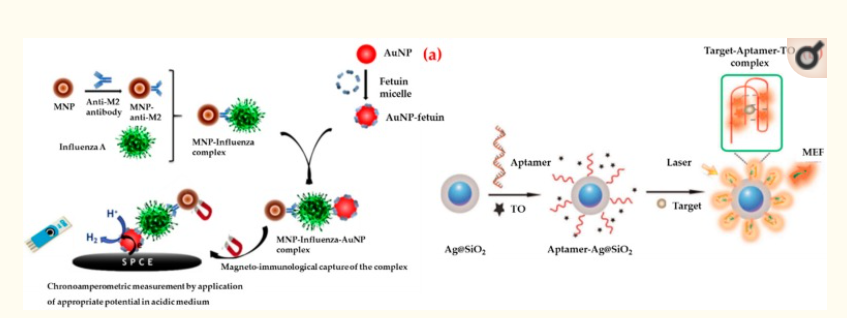
Illustrazione schematica della strategia di sviluppo di biosensori cronoamperometrici basati su nanoparticelle d’oro per il virus dell’influenza (a) e la preparazione di biosensori basati su aptameri e la determinazione della proteina rHA di H5N1 (b). Ripubblicato con il permesso di [115,117].
Inoltre, Tam et al. ha descritto uno studio sull’immobilizzazione del DNA utilizzando nanotubi di carbonio a parete multipla per il rilevamento del virus dell’influenza [116]. Hanno collegato la sonda del DNA al biosensore e hanno caratterizzato l’interazione mediante analisi di spettrometria Raman e infrarossi con trasformata di Fourier. Hanno rilevato l’ibridazione della sonda del DNA e del DNA bersaglio mediante cambiamenti nella conduttanza sulla superficie dei biosensori che portano alla variazione del segnale di uscita del sistema e hanno calcolato il valore limite di rilevamento come 0,5 nM.
Pang et al. ha progettato un biosensore fluorescente per la rilevazione della proteina emoagglutinina ricombinante del virus dell’influenza H5N1 nel siero umano [117]. Come mostrato nella Figura 6b, hanno seguito diversi passaggi per preparare un biosensore fluorescente. In primo luogo, hanno immobilizzato gli aptameri dell’emoagglutinina antiricombinante ricchi di guanina mediante SELEX sulla superficie delle nanoparticelle di biossido di argento e silicio. Quindi, hanno usato l’arancio tiazolo come etichetta fluorescente. Hanno osservato che l’arancio tiazolo era libero senza emissione di fluorescenza in assenza di proteina emoagglutinina ricombinante e la proteina emoagglutinina ricombinante legata al filamento aptamer formava un complesso G-quadruplex stabile quando la proteina emoagglutinina ricombinante veniva aggiunta alla soluzione. Hanno operato la rilevazione della proteina emoagglutinina ricombinante del virus dell’influenza H5N1 sia nel tampone acquoso che nel siero umano con un valore limite di rilevazione di 2 e 3,5 ng/mL.
Inoltre, Volmer et al. ha riportato un metodo di rilevamento ottico del virus dell’influenza A [118]. Hanno osservato che il legame di singoli virioni da discreti cambiamenti nella frequenza di risonanza di una modalità galleria sussurro eccitato in una cavità microsferica. Hanno anche scoperto che l’ampiezza del segnale discreto di spostamento della lunghezza d’onda può essere sufficientemente migliorata riducendo la dimensione della microsfera. Hanno confermato che un meccanismo di rilevamento reattivo con dipendenza inversa dal volume della modalità con nanoparticelle di polistirene delle dimensioni di un virus.
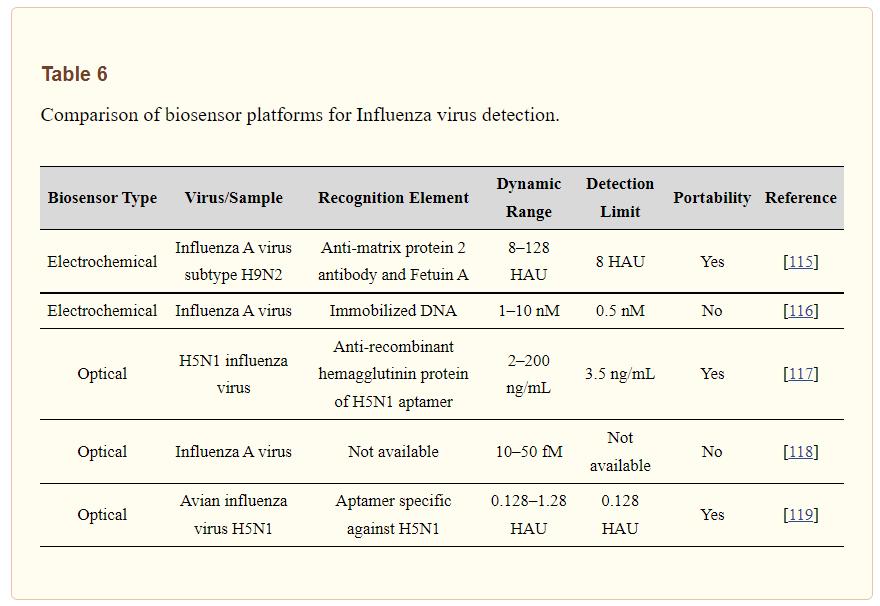
Bai et al. ha preparato un biosensore portatile a risonanza plasmonica di superficie utilizzando un aptamer per il rilevamento del virus dell’influenza aviaria H5N1 in campioni di tampone di pollame [119]. Hanno fabbricato il biosensore utilizzando aptameri selezionati che sono stati biotinilati e quindi immobilizzati sulla superficie in oro del biosensore ricoperta di streptavidina. Gli aptameri immobilizzati hanno catturato l’AIV H5N1 in una soluzione campione, che ha causato un aumento dell’indice di rifrazione. Dopo aver ottimizzato i parametri di streptavidina e aptamer, i risultati hanno mostrato che il valore dell’indice di rifrazione era correlato in modo lineare alla concentrazione di AIV nell’intervallo 0,128-1,28 unità di emoagglutinina (HAU). La tabella 6 fornisce un confronto delle piattaforme di biosensori per il rilevamento del virus dell’influenza
3.7. dengue
La febbre dengue è riemersa come una delle principali sfide per la salute pubblica in tutto il mondo; con 2,5 miliardi di persone a rischio di infezione, sono stati segnalati annualmente più di 100 milioni di casi e 25.000 decessi [120]. Il virus Dengue è un membro del genere Flavivirus di virus a RNA a senso positivo a filamento singolo che causano malattie viscerali e del sistema nervoso centrale negli esseri umani. Le infezioni da virus dengue provocano una malattia inapparente (fino al 75% delle infezioni) o uno spettro di malattie cliniche che vanno dalla febbre dengue autolimitante alla dengue grave, una sindrome emorragica potenzialmente letale e da perdita capillare precedentemente chiamata febbre emorragica dengue e sindrome da shock dengue [121]. Il rilevamento di IgM e glicoproteina dengue NS1 basato su test diagnostici rapidi e metodi ELISA sono i test dengue più utilizzati in molti paesi. Per questo motivo, molti ricercatori hanno approfondito i biosensori come una nuova tecnologia alternativa per il rilevamento del virus della dengue e degli anticorpi della dengue poiché questa tecnica presenta numerosi vantaggi come una maggiore sensibilità, una fabbricazione semplice e conveniente, una possibile miniaturizzazione, un risultato rapido con quantità analisi ed eventuale monitoraggio in loco [122].
Ad esempio, Zhang et al. ha presentato un biosensore a nanofili di silicio per il rilevamento del sierotipo 2 della dengue [123]. Per prima cosa hanno attaccato in modo covalente un acido nucleico peptidico sulla superficie del nanofilo di silicio. Successivamente è stato ottenuto un frammento complementare di sierotipo 2 di dengue e applicato al nanofilo di silicio funzionalizzato con acido nucleico peptidico. Hanno verificato l’ibridazione misurando la variazione di resistenza del biosensore a nanofili di silicio prima e dopo il legame del sierotipo 2 di dengue alla sequenza dell’acido nucleico peptidico (Figura 7a). Hanno riferito che il biosensore a nanofili di silicio può rilevare concentrazioni inferiori a 10 fM degli ampliconi.
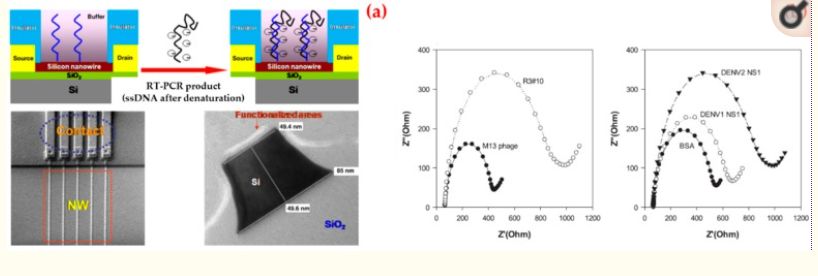
A schematic diagram of the sensor, scanning electron microscope image of nanowire arrays and the corresponding contact lines, transmission electron microscope image of nanowire after surface functionalization (a); selectivity test of selected phage clones: phage particles were incubated with dengue virus NS1 proteins, dengue virus type 1 NS1 and type 2 NS1 protein (b). Republished with permission from [123,124,126].
Lime et al. utilizzato fago polivalente per identificare l’affinità dei peptidi alla proteina NS1 [124]. Hanno mostrato che il peptide era ricco di residui basici dall’analisi della sequenza di amminoacidi. Tra tutti i peptidi testati, il fago selezionato ha mostrato la maggiore diminuzione della corrente nella voltammetria ciclica e un aumento dell’impedenza nella spettroscopia di impedenza elettrochimica dopo il legame con le proteine NS1. Hanno anche rivelato che i cloni fagici erano più specifici nei confronti delle proteine NS1, rispetto all’albumina sierica bovina o al wild type M13 (Figura 7b).
Inoltre, Deng et al. ha sviluppato una piattaforma di rilevamento della membrana di ossido di alluminio anodico per il rilevamento del DNA [125]. Hanno rivestito la membrana di allumina con elettrodi di platino per eliminare la resistenza della soluzione all’esterno dei nanopori e quindi hanno utilizzato la tecnica dell’impedenza elettrochimica per monitorare i cambiamenti di impedenza all’interno dei nanopori dopo il legame al DNA. Hanno dimostrato che la resistenza dei pori aumenta linearmente in risposta alla crescente concentrazione del DNA bersaglio. Inoltre, il biosensore differenzia selettivamente la sequenza complementare da filamenti non corrispondenti a base singola e filamenti non complementari.
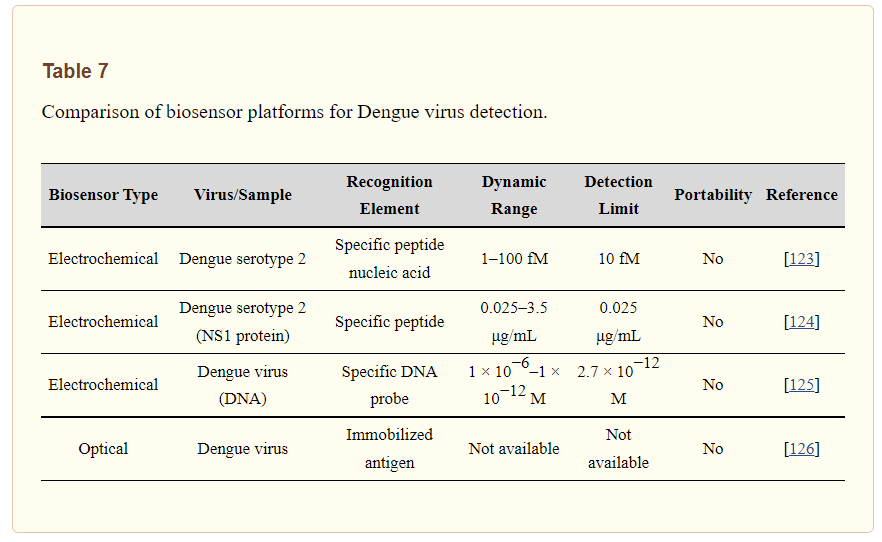
Jahanshahi et al. ha proposto un biosensore di risonanza plasmonica di superficie per la rilevazione del virus anti-dengue in campioni di siero umano [126]. Hanno usato quattro sierotipi del virus dengue come ligandi su un biosensore. Secondo i risultati, hanno mostrato che è necessario un siero di volume di siero ridotto al minimo da un paziente di dengue per indicare la variazione dell’angolo del biosensore di risonanza plasmonica di superficie per determinare il rapporto di ciascun sierotipo di dengue in campioni con elevata sensibilità (83-93%) e specificità (100 %). La tabella 7 fornisce anche un confronto delle piattaforme di biosensori per il rilevamento del virus Dengue.
3.8. Altri virus
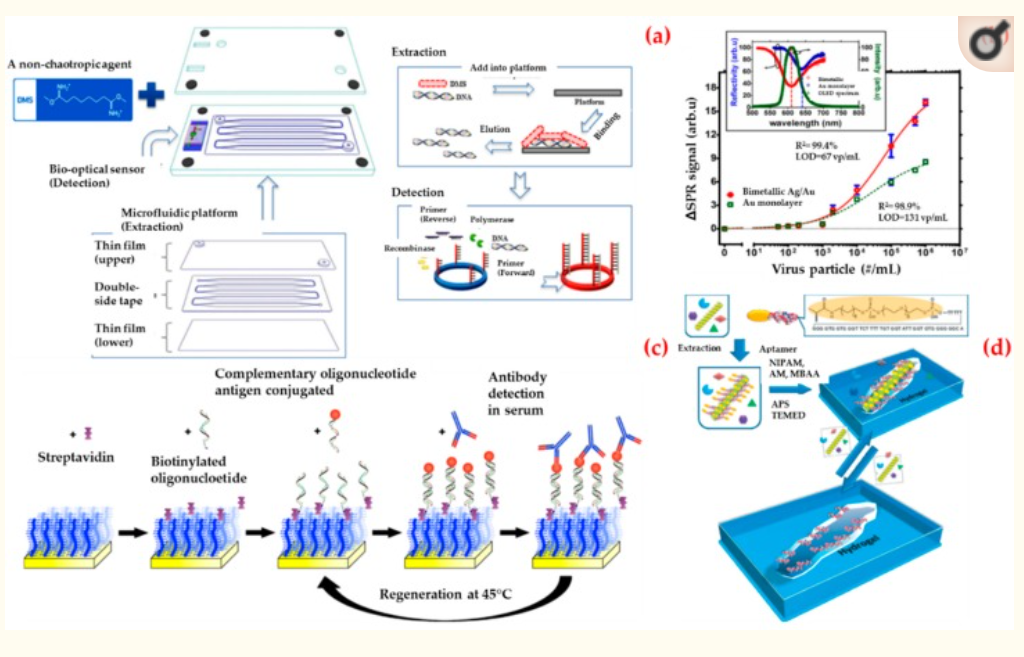
Il rilevamento dei virus è di interesse per una serie di campi tra cui la biomedicina, le scienze ambientali e la biosicurezza [127]. Oltre alle costose apparecchiature e al personale esperto, sono di particolare interesse i sistemi in cui i risultati possono essere letti ad occhio nudo [128,129]. Jin et al. hanno riportato un sistema diagnostico di virus basato su un biosensore ottico e l’elaborazione di campioni microfluidici per il rilevamento di adenovirus umani (Figura 8a) [130]. Hanno prima ottenuto il DNA virale da campioni di adenovirus umano utilizzando una tecnica di estrazione diversa e poi hanno osservato che il biosensore ottico può rilevare dieci copie di adenovirus umano in campioni clinici in mezz’ora. Infine, hanno convalidato l’utilità clinica del sistema diagnostico del virus in tredici campioni umani (dieci dei quali con adenovirus umano e tre con un altro patogeno). Hanno affermato che il sistema di diagnostica dei virus offre una piattaforma diagnostica rapida e sensibile per l’analisi del DNA virale a basso costo, semplicità, tempi di analisi brevi e senza la necessità di strumenti complessi.
Prabowo et al. ha presentato un metodo di rilevamento senza etichetta e di quantificazione rapida per l’enterovirus umano 71 utilizzando un sistema portatile di risonanza plasmonica di superficie [131]. Hanno selezionato la principale proteina del capside dell’enterovirus umano 71 come biomarcatore. Hanno ridotto il tempo sperimentale richiesto per la quantificazione dell’enterovirus umano 71 da sei giorni a diversi minuti. Come illustrato nella Figura 8b, hanno anche stabilito un valore limite di rilevamento di circa 67 particelle virali per millilitro (vp/mL) e, infine, hanno ottenuto un valore limite di rilevamento della principale proteina del capside dell’enterovirus umano rilevamento nel biosensore di risonanza plasmonica di superficie come 4,8 pg/ml.
Riedel et al. ha introdotto una piattaforma di biosensori basata sul sistema di risonanza plasmonica di superficie per la diagnosi di diverse fasi delle infezioni da virus di Epstein-Barr in campioni di siero clinico [132]. Ciò è stato ottenuto mediante il rilevamento simultaneo degli anticorpi contro tre diversi antigeni presenti nel virus e quindi il biosensore è stato attaccato tramite l’ibridazione di oligonucleotidi complementari (Figura 8c). In questo modo, hanno usato ripetutamente la stessa superficie di rilevamento. Hanno affermato che questo approccio servirà come strategia prototipo per lo sviluppo di biosensori per applicazioni mediche.
Bai et al. ha sviluppato un metodo di doppia imprinting basato su un idrogel impresso da virus in un sensore utilizzando tecniche di litografia di impronta (Figura 8d) [133]. Hanno usato un semplice apparato di trasmissione laser per misurare la diffrazione e leggere ad occhio nudo per rilevare il virus della vaiolatura del gambo di mela a concentrazioni fino a (10 ng/mL). Hanno riferito che il limite del valore di rilevamento di questo sistema era inferiore rispetto ad altri metodi commerciali di legame dell’antigene.
nan et al. ha preparato un dispositivo di filtro microfluidico per rilevare e quantificare gli anticorpi del virus del papilloma umano 16 E7 dal sangue intero come tecnologia di assistenza non invasiva per la diagnosi dei tumori maligni associati al virus del papilloma umano [134]. Hanno rilevato l’anticorpo E7 del virus del papilloma umano 16 fino a 2,87 ng/mL. Hanno inoltre convalidato la loro piattaforma in campioni clinici di pazienti e fornito risposte significativamente elevate rispetto ai campioni di controllo. Hanno riferito che questa piattaforma può essere potenzialmente implementata come strumento di pre-test per identificare gruppi ad alto rischio per un ampio monitoraggio dei tumori associati al virus del papilloma umano in contesti con risorse limitate.
Birnbauer et al. ha riportato uno studio sulla preparazione di un sistema di biosensori per l’analisi totale micro per monitorare continuamente la contaminazione virale. Hanno combinato microfluidica contenente polimero nativo e stampato integrato con biosensori dielettrici senza contatto per il rilevamento del sierotipo 2 di Rhinovirus umano. Hanno dimostrato che il legame virale e di dissociazione può essere facilmente rilevato per frequenze specifiche. Hanno completato la rimozione del virus per dimostrare la riutilizzabilità del biosensore a seguito di un aumento di cinquanta volte [135].
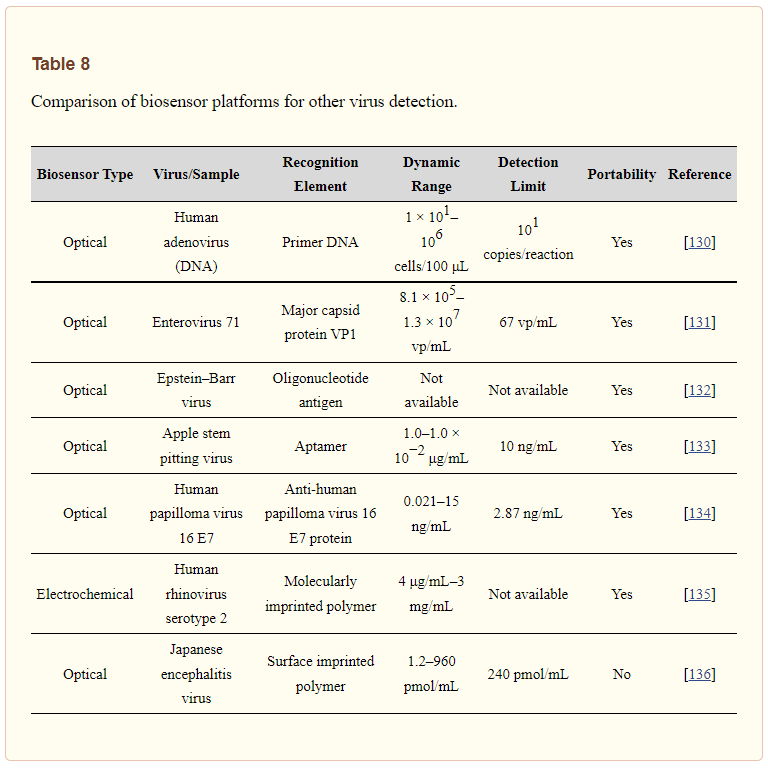
Feng et al. ha studiato il rilevamento fluorescente del virus dell’encefalite giapponese mediante biosensore a base di polimeri impressi molecolarmente sulla superficie. Hanno prima modificato le microsfere di silice con dansil cloruro e poi hanno polimerizzato utilizzando il monomero disponibile e la miscela di reticolanti con il virus dell’encefalite giapponese in condizioni lievi. Dopo la polimerizzazione, hanno eseguito esperimenti di rilevamento e hanno dimostrato che questo biosensore può riconoscere selettivamente il virus dell’encefalite giapponese in presenza di altri agenti competitivi (virus dell’epatite A, virus Simian 40 e virus della rabbia) e rilevare sensibilmente con un livello limite di rilevamento basso (pM). [136]. Infine, la Tabella 8 mostra un confronto tra piattaforme di biosensori per altri rilevamenti di virus.
- Conclusioni
Nella diagnosi medica, i test per biomarcatori specifici vengono eseguiti in laboratori centralizzati utilizzando grandi analizzatori clinici automatizzati che sono generalmente basati su DNA o microarray di proteine, compresi i metodi immunologici tradizionali (saggio radioimmunologico o saggio immunoenzimatico) che richiedono l’uso di etichette. Di solito consentono il rilevamento multiplex di diversi analiti, ma richiedono personale qualificato, molto tempo e molto impegno. D’altro canto, il rapido sviluppo della nanotecnologia e della biotecnologia ha chiaramente migliorato la progettazione e la fabbricazione di nuovi dispositivi per il biosensore nelle applicazioni mediche. I biosensori sono piattaforme desiderabili che presentano numerosi vantaggi, tra cui un’elevata capacità di rilevamento, stabilità, semplicità, affidabilità e convenienza, e possono essere progettati senza influire negativamente sulla sensibilità e sulla riproducibilità degli standard nell’analisi clinica. La fattibilità del biosensore sembra iniziare a lasciare la fase di proof-of-concept e un numero crescente di analiti è già stato rilevato, diverse biomolecole tra cui proteine, ormoni e acidi nucleici, e anche molecole più complesse come esosomi, batteri, virus o cellule che dimostrare la versatilità della biotecnologia. Inoltre, ci sono alcuni esempi di biosensori portatili per determinare alcuni risultati (livelli di glucosio nel sangue o coagulazione del sangue) per l’autotest del paziente. Sebbene siano state sviluppate molte diverse piattaforme di biosensori in questo concetto, non è stato ancora stabilito un chiaro leader nella pratica clinica di routine, ma la diagnostica medica mostra un enorme campo di ricerca che deve ancora affrontare molte sfide irrisolte necessarie per lo sviluppo e la commercializzazione dei dispositivi.
In questa recensione, i recenti sviluppi dei biosensori sono stati ampiamente esaminati per il rilevamento di diversi virus nelle applicazioni mediche. Rispetto alle tecniche convenzionali, queste piattaforme di biosensori mostrano applicazioni più promettenti per migliorare la salute umana per molti paesi e aree rurali delle città.
Vai a:
- Prospettive future
Un potente biosensore dovrebbe avere proprietà intuitive insieme a componenti ad alte prestazioni. Queste importanti caratteristiche sono state adattate per varie applicazioni specifiche come la diagnosi medica correlata alle infezioni. Inoltre, è difficile trovare una piattaforma di biosensori adeguata per le malattie microbiche nel mercato medico a causa dello studio di batteri o profili di marcatori correlati ai virus. Fino a quando questi profili non saranno stabiliti, le piattaforme di biosensori dovrebbero essere mantenute adattabili. I test per i biomarcatori vengono solitamente eseguiti in laboratori con analizzatori automatizzati. La maggior parte di essi si basa su metodi di microarray o immunodosaggio. Oltre agli svantaggi di questi metodi, una piattaforma portatile fornisce risultati corretti in breve tempo. Abbiamo dovuto aspettare molto tempo per scoprire la causa di alcune malattie infettive. Ci sono ancora alcune sfide da superare e alcuni progetti di biosensori portatili sono apparsi di recente a livello di ricerca. Tuttavia, la progressione è piuttosto lenta, un progresso significativo nella tecnologia degli smartphone come diagnostica sanitaria mobile, in particolare, per l’implementazione nei paesi in via di sviluppo e in contesti decentralizzati a basse risorse. La crescita esponenziale nello sviluppo di applicazioni mobili e l’accessibilità economica di queste piattaforme sono chiamate a rivoluzionare l’assistenza sanitaria e ad aprire le porte a una nuova fase nell’accesso alla salute globale.
References
1. Alvarez M.M., Aizenberg J., Analoui M., Andrews A.M., Bisker G., Boyden E.S., Kamm R.D., Karp J.M., Mooney D.J., Oklu R., et al. Emerging trends in micro-and nanoscale technologies in medicine: From basic discoveries to translation. ACS Nano. 2017;11:5195–5214. doi: 10.1021/acsnano.7b01493. [PubMed] [CrossRef] [Google Scholar]2. Malik A.A., Nantasenamat C., Piacham T. Molecularly imprinted polymer for human viral pathogen detection. Mater. Sci. Eng. C. 2017;77:1341–1348. doi: 10.1016/j.msec.2017.03.209. [PubMed] [CrossRef] [Google Scholar]3. Hotez P.J. Blue marble health and “the big three diseases”: HIV/AIDS, tuberculosis, and malaria. Microbes Infect. 2015;17:539–541. doi: 10.1016/j.micinf.2015.05.004. [PubMed] [CrossRef] [Google Scholar]4. Chan Y.K., Gack M.U. Viral evasion of intracellular DNA and RNA sensing. Nat. Rev. Microbiol. 2016;14:360–373. doi: 10.1038/nrmicro.2016.45. [PMC free article] [PubMed] [CrossRef] [Google Scholar]5. Afzal A., Mujahid A., Schirhagl R., Bajwa S., Latif U., Feroz S. Gravimetric viral diagnostics: QCM based biosensors for early detection of viruses. Chemosensors. 2017;5:7. doi: 10.3390/chemosensors5010007. [CrossRef] [Google Scholar]6. Payungporn S., Chutinimitkul S., Chaisingh A., Damrongwantanapokin S., Buranathai C., Amonsin A., Theamboonlers A., Poovorawan Y. Single step multiplex real-time RT-PCR for H5N1 influenza A virus detection. J. Virol. Methods. 2006;131:143–147. doi: 10.1016/j.jviromet.2005.08.004. [PubMed] [CrossRef] [Google Scholar]7. Viswanathan R., George S., Murhekar M.V., Abraham A.M., Singh M.P., Jadhav S.M., Nag V., Naik S., Raut C., Munivenkatappa A., et al. Comparison of two commercial ELISA kits for detection of rubella specific IgM in suspected congenital rubella syndrome cases and rubella IgG antibodies in a serosurvey of pregnant women: Comparison of two commercial ELISA kits for detection of rubella specific IgM and IgG antibodies. Diagn. Microbiol. Infect. Dis. 2019 in press. [PubMed] [Google Scholar]8. Gourinat A.C., O’Connor O., Calvez E., Goarant C., Dupont-Rouzeyrol M. Detection of Zika virus in urine. Emerg. Infect. Dis. 2015;21:84–86. doi: 10.3201/eid2101.140894. [PMC free article] [PubMed] [CrossRef] [Google Scholar]9. Krishna V.D., Wu K., Perez A.M., Wang J.P. Giant magnetoresistance-based biosensor for detection of influenza A virus. Front. Microbiol. 2016;7:400–408. doi: 10.3389/fmicb.2016.00400. [PMC free article] [PubMed] [CrossRef] [Google Scholar]10. Navakul K., Warakulwit C., Yenchitsomanus P.T., Panya A., Lieberzeit P.A., Sangma C. A novel method for dengue virus detection and antibody screening using a graphene-polymer based electrochemical biosensor. NBM. 2017;13:549–557. doi: 10.1016/j.nano.2016.08.009. [PubMed] [CrossRef] [Google Scholar]11. Khater M., de la Escosura-Muñiz A., Merkoçi A. Biosensors for plant pathogen detection. Biosens. Bioelectron. 2017;93:72–86. doi: 10.1016/j.bios.2016.09.091. [PubMed] [CrossRef] [Google Scholar]12. Tsang M.K., Ye W., Wang G., Li J., Yang M., Hao J. Ultrasensitive detection of Ebola virus oligonucleotide based on upconversion nanoprobe/nanoporous membrane system. ACS Nano. 2016;10:598–605. doi: 10.1021/acsnano.5b05622. [PubMed] [CrossRef] [Google Scholar]13. Wong C.L., Chua M., Mittman H., Choo L.X., Lim H.Q., Olivo M. A phase-intensity surface plasmon resonance biosensor for avian influenza A (H5N1) detection. Sensors. 2017;17:2363. doi: 10.3390/s17102363. [PMC free article] [PubMed] [CrossRef] [Google Scholar]14. Altintas Z., Gittens M., Pocock J., Tothill I.E. Biosensors for waterborne viruses: Detection and removal. Biochimie. 2015;115:144–154. doi: 10.1016/j.biochi.2015.05.010. [PubMed] [CrossRef] [Google Scholar]15. Justino C.I., Freitas A.C., Pereira R., Duarte A.C., Santos T.A.R. Recent developments in recognition elements for chemical sensors and biosensors. Trends Anal. Chem. 2015;68:2–17. doi: 10.1016/j.trac.2015.03.006. [CrossRef] [Google Scholar]16. Saylan Y., Yılmaz F., Özgür E., Derazshamshir A., Bereli N., Yavuz H., Denizli A. Nanotechnology Characterization Tools for Biosensing and Medical Diagnosis. In: Kumar C.S.S.R., editor. Surface Plasmon Resonance Sensors for Medical Diagnosis. Springer; Berlin/Heidelberg, Germany: 2018. pp. 425–458. [Google Scholar]17. Wang X.D., Wolfbeis O.S. Fiber-optic chemical sensors and biosensors (2013–2015) Anal. Chem. 2015;88:203–227. doi: 10.1021/acs.analchem.5b04298. [PubMed] [CrossRef] [Google Scholar]18. Bharadwaj R., Mukherji S., Mukherji S. Probing the localized surface plasmon field of a gold nanoparticle-based fibre optic biosensor. Plasmonics. 2016;11:753–761. doi: 10.1007/s11468-015-0106-0. [CrossRef] [Google Scholar]19. Saylan Y., Yılmaz F., Derazshamshir A., Yılmaz E., Denizli A. Synthesis of hydrophobic nanoparticles for real-time lysozyme detection using surface plasmon resonance sensor. J. Mol. Recognit. 2017;30:e2631. doi: 10.1002/jmr.2631. [PubMed] [CrossRef] [Google Scholar]20. Eissa S., Siaj M., Zourob M. Aptamer-based competitive electrochemical biosensor for brevetoxin-2. Biosens. Bioelectron. 2015;69:148–154. doi: 10.1016/j.bios.2015.01.055. [PubMed] [CrossRef] [Google Scholar]21. Hu T., Zhang L., Wen W., Zhang X., Wang S. Enzyme catalytic amplification of miRNA-155 detection with graphene quantum dot-based electrochemical biosensor. Biosens. Bioelectron. 2016;77:451–456. doi: 10.1016/j.bios.2015.09.068. [PubMed] [CrossRef] [Google Scholar]22. Shin S.R., Zhang Y.S., Kim D.J., Manbohi A., Avci H., Silvestri A., Aleman J., Hu N., Kilic T., Keung W., et al. Aptamer-based microfluidic electrochemical biosensor for monitoring cell-secreted trace cardiac biomarkers. Anal. Chem. 2016;88:10019–10027. doi: 10.1021/acs.analchem.6b02028. [PMC free article] [PubMed] [CrossRef] [Google Scholar]23. Do Nascimento N.M., Juste-Dolz A., Grau-García E., Román-Ivorra J.A., Puchades R., Maquieira A., Morais S., Gimenez-Romero D. Label-free piezoelectric biosensor for prognosis and diagnosis of Systemic Lupus Erythematosus. Biosens. Bioelectron. 2017;90:166–173. doi: 10.1016/j.bios.2016.11.004. [PubMed] [CrossRef] [Google Scholar]24. Pohanka M. Piezoelectric biosensor for the determination of tumor necrosis factor alpha. Talanta. 2018;178:970–973. doi: 10.1016/j.talanta.2017.10.031. [PubMed] [CrossRef] [Google Scholar]25. Giamblanco N., Conoci S., Russo D., Marletta G. Single-step label-free hepatitis B virus detection by a piezoelectric biosensor. RSC Adv. 2015;5:38152–38158. doi: 10.1039/C5RA03467A. [CrossRef] [Google Scholar]26. Battal D., Akgönüllü S., Yalcin M.S., Yavuz H., Denizli A. Molecularly imprinted polymer based quartz crystal microbalance sensor system for sensitive and label-free detection of synthetic cannabinoids in urine. Biosens. Bioelectron. 2018;111:10–17. doi: 10.1016/j.bios.2018.03.055. [PubMed] [CrossRef] [Google Scholar]27. Zhang Q., Zhang D., Lu Y., Yao Y., Li S., Liu Q. Graphene oxide-based optical biosensor functionalized with peptides for explosive detection. Biosens. Bioelectron. 2015;68:494–499. doi: 10.1016/j.bios.2015.01.040. [PubMed] [CrossRef] [Google Scholar]28. Kabessa Y., Eyal O., Bar-On O., Korouma V., Yagur-Kroll S., Belkin S., Agranat A.J. Standoff detection of explosives and buried landmines using fluorescent bacterial sensor cells. Biosens. Bioelectron. 2016;79:784–788. doi: 10.1016/j.bios.2016.01.011. [PubMed] [CrossRef] [Google Scholar]29. Osman B., Uzun L., Beşirli N., Denizli A. Microcontact imprinted surface plasmon resonance sensor for myoglobin detection. Mater. Sci. Eng. C. 2013;33:3609–3614. doi: 10.1016/j.msec.2013.04.041. [PubMed] [CrossRef] [Google Scholar]30. Wang H.B., Zhang H.D., Chen Y., Liu Y.M. A fluorescent biosensor for protein detection based on poly (thymine)-templated copper nanoparticles and terminal protection of small molecule-linked DNA. Biosens. Bioelectron. 2015;74:581–586. doi: 10.1016/j.bios.2015.07.021. [PubMed] [CrossRef] [Google Scholar]31. Bartold K., Pietrzyk-Le A., Golebiewska K., Lisowski W., Cauteruccio S., Licandro E., D’Souza F., Kutner W. Oligonucleotide determination via peptide nucleic acid macromolecular imprinting in an electropolymerized cg-rich artificial oligomer analogue. ACS Appl. Mater. Int. 2018;10:27562–27569. doi: 10.1021/acsami.8b09296. [PubMed] [CrossRef] [Google Scholar]32. Shoaie N., Forouzandeh M., Omidfar K. Highly sensitive electrochemical biosensor based on polyaniline and gold nanoparticles for DNA detection. IEEE Sens. J. 2018;18:1835–1843. doi: 10.1109/JSEN.2017.2787024. [CrossRef] [Google Scholar]33. Cheng S., Hideshima S., Kuroiwa S., Nakanishi T., Osaka T. Label-free detection of tumor markers using field effect transistor (FET)-based biosensors for lung cancer diagnosis. Sens. Actuators B-Chem. 2015;212:329–334. doi: 10.1016/j.snb.2015.02.038. [CrossRef] [Google Scholar]34. Atay S., Pişkin K., Yılmaz F., Çakır C., Yavuz H., Denizli A. Quartz crystal microbalance based biosensors for detecting highly metastatic breast cancer cells via their transferrin receptors. Anal. Methods. 2016;8:153–162. doi: 10.1039/C5AY02898A. [CrossRef] [Google Scholar]35. Cheeveewattanagul N., Morales-Narváez E., Hassan A.R.H., Bergua J.F., Surareungchai W., Somasundrum M., Merkoçi A. Straightforward immunosensing platform based on graphene oxide-decorated nanopaper: A highly sensitive and fast biosensing approach. Adv. Funct. Mater. 2017;27:1702741–1702749. doi: 10.1002/adfm.201702741. [CrossRef] [Google Scholar]36. Erdem Ö., Saylan Y., Cihangir N., Denizli A. Molecularly imprinted nanoparticles based plasmonic sensors for real-time Enterococcus faecalis detection. Biosens. Biolectron. 2019;126:608–614. doi: 10.1016/j.bios.2018.11.030. [PubMed] [CrossRef] [Google Scholar]37. Anik U., Tepeli Y., Diouani M.F. Fabrication of electrochemical model influenza a virus biosensor based on the measurements of neuroaminidase enzyme activity. Anal. Chem. 2016;88:6151–6153. doi: 10.1021/acs.analchem.6b01720. [PubMed] [CrossRef] [Google Scholar]38. Bäcker M., Koch C., Eiben S., Geiger F., Eber F., Gliemann H., Poghossian A., Wege C., Schöning M.J. Tobacco mosaic virus as enzyme nanocarrier for electrochemical biosensors. Sens. Actuators B-Chem. 2017;238:716–722. doi: 10.1016/j.snb.2016.07.096. [CrossRef] [Google Scholar]39. Dudak F.C., Boyaci İ.H. Peptide-based surface plasmon resonance biosensor for detection of staphylococcal enterotoxin b. Food Anal. Methods. 2014;7:506–511. doi: 10.1007/s12161-013-9739-9. [CrossRef] [Google Scholar]40. Cunha I., Biltes R., Sales M.G.F., Vasconcelos V. Aptamer-based biosensors to detect aquatic phycotoxins and cyanotoxins. Sensors. 2018;18:2367. doi: 10.3390/s18072367. [PMC free article] [PubMed] [CrossRef] [Google Scholar]41. Lv M., Liu Y., Geng J., Kou X., Xin Z., Yang D. Engineering nanomaterials-based biosensors for food safety detection. Biosens. Bioelectron. 2018;106:122–128. doi: 10.1016/j.bios.2018.01.049. [PubMed] [CrossRef] [Google Scholar]42. Erdem Ö., Saylan Y., Andaç M., Denizli A. Molecularly imprinted polymers for removal of metal ions: An alternative treatment method. Biomimetics. 2018;3:38. doi: 10.3390/biomimetics3040038. [PMC free article] [PubMed] [CrossRef] [Google Scholar]43. Dibekkaya H., Saylan Y., Yılmaz F., Derazshamshir A., Denizli A. Surface plasmon resonance sensors for real-time detection of cyclic citrullinated peptide antibodies. J. Macromol. Sci. A. 2016;53:585–594. doi: 10.1080/10601325.2016.1201756. [CrossRef] [Google Scholar]44. Saylan Y., Denizli A. Molecular fingerprints of hemoglobin on a nanofilm chip. Sensors. 2018;18:3016. doi: 10.3390/s18093016. [PMC free article] [PubMed] [CrossRef] [Google Scholar]45. Mao C., Liu A., Cao B. Virus-based chemical and biological sensing. Angew. Chem. Int. Ed. 2009;48:6790–6810. doi: 10.1002/anie.200900231. [PMC free article] [PubMed] [CrossRef] [Google Scholar]46. Goode J.A., Rushworth J.V.H., Millner P.A. Biosensor regeneration: A review of common techniques and outcomes. Langmuir. 2015;31:6267–6276. doi: 10.1021/la503533g. [PubMed] [CrossRef] [Google Scholar]47. Verma N., Bhardwaj A. Biosensor technology for pesticides—A review. Biotechnol. Appl. Biochem. 2015;175:3093–3119. doi: 10.1007/s12010-015-1489-2. [PubMed] [CrossRef] [Google Scholar]48. Saylan Y., Yılmaz F., Özgür E., Derazshamshir A., Yavuz H., Denizli A. Molecularly imprinting of macromolecules for sensors applications. Sensors. 2017;17:898. doi: 10.3390/s17040898. [PMC free article] [PubMed] [CrossRef] [Google Scholar]49. Cho K.H., Shin D.H., Oh J., An J.H., Lee J.S., Jang J. Multidimensional conductive nanofilm-based flexible aptasensor for ultrasensitive and selective HBsAg detection. ACS Appl. Mater. Interfaces. 2018;10:28412–28419. doi: 10.1021/acsami.8b09918. [PubMed] [CrossRef] [Google Scholar]50. La Spada L., Vegni L. Electromagnetic nanoparticles for sensing and medical diagnostic applications. Materials. 2018;11:603. doi: 10.3390/ma11040603. [PMC free article] [PubMed] [CrossRef] [Google Scholar]51. Pang Y., Jian J., Tu T., Yang Z., Ling J., Li Y., Wang X., Qiao Y., Tian H., Yang Y., et al. Wearable humidity sensor based on porous graphene network for respiration monitoring. Biosens. Bioelectron. 2018;116:123–129. doi: 10.1016/j.bios.2018.05.038. [PubMed] [CrossRef] [Google Scholar]52. Russo L., Leva Bueno J., Bergua J.F., Costantini M., Giannetto M., Puntes V., de la Escosura-Muñiz A., Merkoci A. Low-cost strategy for the development of a rapid electrochemical assay for bacteria detection based on AuAg nanoshells. ACS Omega. 2018;3:18849–18856. doi: 10.1021/acsomega.8b02458. [CrossRef] [Google Scholar]53. Thévenot D.R., Toth K., Durst R.A., Wilson G.S. Electrochemical biosensors: Recommended definitions and classification. Anal. Lett. 2001;34:635–659. doi: 10.1081/AL-100103209. [CrossRef] [Google Scholar]54. Zhu J., Gan H., Wu J., Ju H. Molecular machine powered surface programmatic chain reaction for highly sensitive electrochemical detection of protein. Anal. Chem. 2018;90:5503–5508. doi: 10.1021/acs.analchem.8b01217. [PubMed] [CrossRef] [Google Scholar]55. Rauf S., Mishra G.K., Azhar J., Mishra R.K., Goud K.Y., Nawaz M.A.H., Martyd J.L., Hayat A. Carboxylic group riched graphene oxide based disposable electrochemical immunosensor for cancer biomarker detection. Anal. Biochem. 2018;545:13–19. doi: 10.1016/j.ab.2018.01.007. [PubMed] [CrossRef] [Google Scholar]56. Kuralay F., Dükar N., Bayramlı Y. Poly-l-lysine coated surfaces for ultrasensitive nucleic acid detection. Electroanalysis. 2018;30:1556–1565. doi: 10.1002/elan.201800153. [CrossRef] [Google Scholar]57. Saylan Y., Akgönüllü S., Yavuz H., Ünal S., Denizli A. Molecularly imprinted polymer based sensors for medical applications. Sensors. 2019;19:1279. doi: 10.3390/s19061279. [PMC free article] [PubMed] [CrossRef] [Google Scholar]58. Yu X., Chen F., Wang R., Li Y. Whole-bacterium SELEX of DNA aptamers for rapid detection of E. coli O157:H7 using a QCM sensor. J. Biotechnol. 2018;266:39–49. doi: 10.1016/j.jbiotec.2017.12.011. [PubMed] [CrossRef] [Google Scholar]59. Özgür E., Yılmaz E., Şener G., Uzun L., Say R., Denizli A. A new molecular imprinting-based mass-sensitive sensor for real-time detection of 17β-estradiol from aqueous solution. Environ. Prog. Sustain. 2013;32:1164–1169. doi: 10.1002/ep.11718. [CrossRef] [Google Scholar]60. Bunroddith K., Viseshakul N., Chansiri K., Lieberzeit P. QCM-based rapid detection of PCR amplification products of Ehrlichia canis. Anal. Chim. Acta. 2018;1001:106–111. doi: 10.1016/j.aca.2017.10.037. [PubMed] [CrossRef] [Google Scholar]61. Suri C.R. Advances in Biosensors, Perpectives in Biosensors. Volume 5. Elsevier Science; Amsterdam, The Netherlands: 2003. Immunosensors for pesticide monitoring; pp. 161–176. [Google Scholar]62. Sciacca B., François A., Hoffmann P., Monro T.M. Multiplexing of radiative-surface plasmon resonance for the detection of gastric cancer biomarkers in a single optical fiber. Sens. Actuators B-Chem. 2013;183:454–458. doi: 10.1016/j.snb.2013.03.131. [CrossRef] [Google Scholar]63. Saini A., Kaur N., Singh N. A highly fluorescent sensor based on hybrid nanoparticles for selective determination of furosemide in aqueous medium. Sens. Actuators B Chem. 2016;228:221–230. doi: 10.1016/j.snb.2016.01.026. [CrossRef] [Google Scholar]64. Chang J.E., Lee D.S., Ban S.W., Oh J., Jung M.Y., Kim S.H., Park S.J., Persaud K., Jheon S. Analysis of volatile organic compounds in exhaled breath for lung cancer diagnosis using a sensor system. Sens. Actuators B-Chem. 2018;255:800–807. doi: 10.1016/j.snb.2017.08.057. [CrossRef] [Google Scholar]65. Saylan Y., Akgönüllü S., Çimen D., Derazshamshir A., Bereli N., Yılmaz F., Denizli A. Surface plasmon resonance nanosensors based on molecularly imprinted nanofilm for detection of pesticides. Sens. Actuators B Chem. 2017;241:446–454. doi: 10.1016/j.snb.2016.10.017. [CrossRef] [Google Scholar]66. Choi D.H., Thaxton A., Cheol Jeong I., Kim K., Sosnay P.R., Cutting G.R., Searson P.C. Sweat test for cystic fibrosis: Wearable sweat sensor vs. standard laboratory test. J. Cyst. Fibros. 2018;17:e35–e38. [PubMed] [Google Scholar]67. Justino C.I.L., Rocha-Santos T.A.P., Cardoso S., Duarte A.C. Strategies for enhancing the analytical performance of nanomaterial-based sensors. Trends Anal. Chem. 2013;47:27–36. doi: 10.1016/j.trac.2013.02.004. [CrossRef] [Google Scholar]68. Loyez M., Larrieu J.C., Chevineau S., Remmelink M., Leduc C., Bondue B., Lambert P., Devière J., Wattiez R., Caucheteur C. In situ cancer diagnosis through online plasmonics. Biosens. Bioelectron. 2019;131:104–112. doi: 10.1016/j.bios.2019.01.062. [PubMed] [CrossRef] [Google Scholar]69. Theint H.T., Walsh J.E., Wong S.T., Von K., Shitan M. Development of an optical biosensor for the detection of Trypanosoma evansi and Plasmodium berghei. Spectrochim. Acta A. 2019;218:348–358. doi: 10.1016/j.saa.2019.04.008. [PubMed] [CrossRef] [Google Scholar]70. Abolhasan R., Mehdizadeh A., Rashidi M.R., Aghebati-Maleki L., Yousefi M. Application of hairpin DNA-based biosensors with various signal amplification strategies in clinical diagnosis. Biosens. Bioelectron. 2019;129:164–174. doi: 10.1016/j.bios.2019.01.008. [PubMed] [CrossRef] [Google Scholar]71. Dükar N., Tunç S., Öztürk K., Demirci S., Dumangöz M., Sönmez Çelebi M., Kuralay F. Highly sensitive and selective dopamine sensing in biological fluids with one-pot prepared graphene/poly(o-phenylenediamine) modified electrodes. Mater. Chem. Phys. 2019;228:357–362. doi: 10.1016/j.matchemphys.2019.02.043. [CrossRef] [Google Scholar]72. Jenik M., Schirhagl R., Schirk C., Hayden O., Lieberzeit P., Blaas D., Paul G., Dickert F.L. Sensing picornaviruses using molecular imprinting techniques on a quartz crystal microbalance. Anal. Chem. 2009;81:5320–5326. doi: 10.1021/ac8019569. [PubMed] [CrossRef] [Google Scholar]73. Schirhagl R., Lieberzeit P.A., Dickert F.L. Chemosensors for viruses based on artificial immunoglobulin copies. Adv. Mater. 2010;22:2078–2081. doi: 10.1002/adma.200903517. [PubMed] [CrossRef] [Google Scholar]74. Cheng D., Yu M., Fu F., Han W., Li G., Xie J., Song Y., Swihart M.T., Song E. Dual recognition strategy for specific and sensitive detection of bacteria using aptamer-coated magnetic beads and antibiotic-capped gold nanoclusters. Anal. Chem. 2016;88:820–825. doi: 10.1021/acs.analchem.5b03320. [PubMed] [CrossRef] [Google Scholar]75. Liu X., Zhang C., Liu K., Wang H., Lu C., Li H., Hua K., Zhu K., Hui W., Cui Y., et al. Multiple SNPs detection based on lateral flow assay for phenylketonuria diagnostic. Anal. Chem. 2018;90:3430–3436. doi: 10.1021/acs.analchem.7b05113. [PubMed] [CrossRef] [Google Scholar]76. Zhang Z., Sohgawa M., Yamashita K., Noda M. A micromechanical cantilever-based liposome biosensor for characterization of protein-membrane interaction. Electroanalysis. 2016;28:620–625. doi: 10.1002/elan.201500412. [CrossRef] [Google Scholar]77. Tardivo M., Toffoli V., Fracasso G., Borin D., Dal Zilio S., Colusso A., Carrato S., Scoles G., Meneghettie M., Colombatti M., et al. Parallel optical read-out of micromechanical pillars applied to prostate specific membrane antigen detection. Biosens. Bioelectron. 2015;72:393–399. doi: 10.1016/j.bios.2015.05.026. [PubMed] [CrossRef] [Google Scholar]78. Casadio S., Lowdon J.W., Betlem K., Ueta J.T., Foster C.W., Cleij T.J., van Grinsven B., Sutcliffe O.B., Banks C.E., Peeters M. Development of a novel flexible polymer-based biosensor platform for the thermal detection of noradrenaline in aqueous solutions. Chem. Eng. J. 2017;315:459–468. doi: 10.1016/j.cej.2017.01.050. [CrossRef] [Google Scholar]79. Wang Z., Jinlong L., An Z., Kimura M., Ono T. Enzyme immobilization in completely packaged freestanding SU-8 microfluidic channel by electro click chemistry for compact thermal biosensor. Process Biochem. 2019;79:57–64. doi: 10.1016/j.procbio.2018.12.007. [CrossRef] [Google Scholar]80. Khan M.S., Misra S.K., Dighe K., Wang Z., Schwartz-Duval A.S., Sar D., Pan D. Electrically-receptive and thermally-responsive paper-based sensor chip for rapid detection of bacterial cells. Biosens. Bioelectron. 2018;110:132–140. doi: 10.1016/j.bios.2018.03.044. [PubMed] [CrossRef] [Google Scholar]81. van Grinsven B., Eersels K., Akkermans O., Ellermann S., Kordek A., Peeters M., Deschaume O., Bartic C., Dilien H., Redeker E.S., et al. Label-free detection of Escherichia coli based on thermal transport through surface imprinted polymers. ACS Sens. 2016;1:1140–1147. doi: 10.1021/acssensors.6b00435. [CrossRef] [Google Scholar]82. Inci F., Tokel O., Wang S., Gurkan U.A., Tasoglu S., Kuritzkes D.R., Demirci U. Nanoplasmonic quantitative detection of intact viruses from unprocessed whole blood. ACS Nano. 2013;7:4733–4745. doi: 10.1021/nn3036232. [PMC free article] [PubMed] [CrossRef] [Google Scholar]83. HIV/AIDS. [(accessed on 1 February 2019)]; Available online: https://www.who.int/en/news-room/fact-sheets/detail/hiv-aids.84. Babamiri B., Salimi A., Hallaj R. A molecularly imprinted electrochemiluminescence sensor for ultrasensitive HIV-1 gene detection using EuS nanocrystals as luminophore. Biosens. Bioelectron. 2018;117:332–339. doi: 10.1016/j.bios.2018.06.003. [PubMed] [CrossRef] [Google Scholar]85. Lu C.H., Zhang Y., Tang S.F., Fang Z.B., Yang H.H., Chen X., Chen G.N. Sensing HIV related protein using epitope imprinted hydrophilic polymer coated quartz crystal microbalance. Biosens. Bioelectron. 2012;31:439–444. doi: 10.1016/j.bios.2011.11.008. [PubMed] [CrossRef] [Google Scholar]86. Shafiee H., Lidstone E.A., Jahangir M., Inci F., Hanhauser E., Henrich T.J., Kuritzkes D.R., Cunningham B.T., Demirci U. Nanostructured optical photonic crystal biosensor for HIV viral load measurement. Sci. Rep. 2014;4:4116–4123. doi: 10.1038/srep04116. [PMC free article] [PubMed] [CrossRef] [Google Scholar]87. Seeger C., Mason W.S. Molecular biology of hepatitis B virus infection. Virology. 2015;479:672–686. doi: 10.1016/j.virol.2015.02.031. [PMC free article] [PubMed] [CrossRef] [Google Scholar]88. Lavanchy D., Kane M. Hepatitis B Virus in Human Diseases. Humana Press; Cham, Switzerland: 2016. Global epidemiology of hepatitis B virus infection; pp. 187–203. [Google Scholar]89. Hassen W.M., Chaix C., Abdelghani A., Bessueille F., Leonard D., Jaffrezic-Renault N. An impedimetric DNA sensor based on functionalized magnetic nanoparticles for HIV and HBV detection. Sens. Actuators B-Chem. 2008;134:755–760. doi: 10.1016/j.snb.2008.06.020. [CrossRef] [Google Scholar]90. Tam Y.J., Zeenathul N.A., Rezaei M.A., Mustafa N.H., Azmi M.L.M., Bahaman A.R., Lo S.C., Tan J.S., Hani H., Rasedee A. Wide dynamic range of surface-plasmon-resonance-based assay for hepatitis B surface antigen antibody optimal detection in comparison with ELISA. Biotechnol. Appl. Biochem. 2017;64:735–744. doi: 10.1002/bab.1528. [PubMed] [CrossRef] [Google Scholar]91. Uzun L., Say R., Ünal S., Denizli A. Production of surface plasmon resonance based assay kit for hepatitis diagnosis. Biosens. Bioelectron. 2009;24:2878–2884. doi: 10.1016/j.bios.2009.02.021. [PubMed] [CrossRef] [Google Scholar]92. Li X., Scida K., Crooks R.M. Detection of hepatitis B virus DNA with a paper electrochemical sensor. Anal. Chem. 2015;87:9009–9015. doi: 10.1021/acs.analchem.5b02210. [PubMed] [CrossRef] [Google Scholar]93. İstek M.M., Erdem M.M., Gürsan A.E. Impedimetric nanobiosensor for the detection of sequence-selective DNA hybridization. Hacet. J. Biol. Chem. 2019;46:495–503. [Google Scholar]94. Zengin A., Tamer U., Caykara T. SERS detection of hepatitis B virus DNA in a temperature-responsive sandwich hybridization assay. J. Raman Spectrosc. 2017;48:668–672. doi: 10.1002/jrs.5109. [CrossRef] [Google Scholar]95. Liu Y., Shen T., Hu L., Gong H., Chen C., Chen X., Cai C. Development of a thermosensitive molecularly imprinted polymer resonance light scattering sensor for rapid and highly selective detection of hepatitis A virus in vitro. Sens. Actuators B-Chem. 2017;253:1188–1193. doi: 10.1016/j.snb.2017.07.166. [CrossRef] [Google Scholar]96. Qiu X., Wong G., Audet J., Bello A., Fernando L., Alimonti J.B., Fausther-Bovendo H., Wei H., Aviles J., Hiatt E., et al. Reversion of advanced Ebola virus disease in nonhuman primates with ZMapp. Nature. 2014;514:47–62. doi: 10.1038/nature13777. [PMC free article] [PubMed] [CrossRef] [Google Scholar]97. Kreil T.R. Treatment of Ebola virus infection with antibodies from reconvalescent donors. Emerg. Infect. Dis. 2015;21:521–523. doi: 10.3201/eid2103.141838. [PMC free article] [PubMed] [CrossRef] [Google Scholar]98. Baca J.T., Severns V., Lovato D., Branch D.W., Larson R.S. Rapid detection of Ebola virus with a reagent-free, point-of-care biosensor. Sensors. 2015;15:8605–8614. doi: 10.3390/s150408605. [PMC free article] [PubMed] [CrossRef] [Google Scholar]99. Ilkhani H., Farhad S. A novel electrochemical DNA biosensor for Ebola virus detection. Anal. Biochem. 2018;557:151–155. doi: 10.1016/j.ab.2018.06.010. [PubMed] [CrossRef] [Google Scholar]100. Yanik A.A., Huang M., Kamohara O., Artar A., Geisbert T.W., Connor J.H., Altug H. An optofluidic nanoplasmonic biosensor for direct detection of live viruses from biological media. Nano Lett. 2010;10:4962–4969. doi: 10.1021/nl103025u. [PMC free article] [PubMed] [CrossRef] [Google Scholar]101. Cai H., Parks J.W., Wall T.A., Stott M.A., Stambaugh A., Alfson K., Griffiths A., Mathies R.A., Carrion R., Patterson J.L., et al. Optofluidic analysis system for amplification-free, direct detection of Ebola infection. Sci. Rep. 2015;5:14494–14502. doi: 10.1038/srep14494. [PMC free article] [PubMed] [CrossRef] [Google Scholar]102. Hennessey M., Fischer M., Staples J.E. Zika virus spreads to new areas—Region of the Americas, May 2015–January 2016. Am. J. Transplant. 2016;16:1031–1034. doi: 10.1111/ajt.13743. [PubMed] [CrossRef] [Google Scholar]103. Meagher R.J., Negrete O.A., Van Rompay K.K. Engineering paper-based sensors for Zika virus. Trends Mol. Med. 2016;22:529–530. doi: 10.1016/j.molmed.2016.05.009. [PubMed] [CrossRef] [Google Scholar]104. Afsahi S., Lerner M.B., Goldstein J.M., Lee J., Tang X., Bagarozzi D.A., Jr., Pan D., Locascio L., Walker A., Barron F., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosens. Bioelectron. 2018;100:85–88. doi: 10.1016/j.bios.2017.08.051. [PubMed] [CrossRef] [Google Scholar]105. Kaushik A., Yndart A., Kumar S., Jayant R.D., Vashist A., Brown A.N., Li C., Nair M. A sensitive electrochemical immunosensor for label-free detection of Zika-virus protein. Sci. Rep. 2018;8:9700–9705. doi: 10.1038/s41598-018-28035-3. [PMC free article] [PubMed] [CrossRef] [Google Scholar]106. Song J., Mauk M.G., Hackett B.A., Cherry S., Bau H.H., Liu C. Instrument-free point-of-care molecular detection of Zika virus. Anal. Chem. 2016;88:7289–7294. doi: 10.1021/acs.analchem.6b01632. [PMC free article] [PubMed] [CrossRef] [Google Scholar]107. Robilotti E., Deresinski S., Pinsky B.A. Norovirus. Clin. Microbiol. Rev. 2015;28:134–164. doi: 10.1128/CMR.00075-14. [PMC free article] [PubMed] [CrossRef] [Google Scholar]108. Hwang H.J., Ryu M.Y., Park C.Y., Ahn J., Park H.G., Choi C., Ha S.D., Park T.J., Park J.P. High sensitive and selective electrochemical biosensor: Label-free detection of human norovirus using affinity peptide as molecular binder. Biosens. Bioelectron. 2017;87:164–170. doi: 10.1016/j.bios.2016.08.031. [PubMed] [CrossRef] [Google Scholar]109. Bierhoff M., Arvelo W., Estevez A., Bryan J., McCracken J.P., López M.R., López B., Parashar U.D., Lindblade K.A., Hall A.J. Incidence and clinical profile of norovirus disease in Guatemala, 2008–2013. Clin. Infect. Dis. 2018;67:430–436. doi: 10.1093/cid/ciy091. [PubMed] [CrossRef] [Google Scholar]110. Ashiba H., Sugiyama Y., Wang X., Shirato H., Higo-Moriguchi K., Taniguchi K., Ohki Y., Fujimaki M. Detection of norovirus virus-like particles using a surface plasmon resonance-assisted fluoroimmunosensor optimized for quantum dot fluorescent labels. Biosens. Bioelectron. 2017;93:260–266. doi: 10.1016/j.bios.2016.08.099. [PubMed] [CrossRef] [Google Scholar]111. Lee J., Morita M., Takemura K., Park E.Y. A multi-functional gold/iron-oxide nanoparticle-CNT hybrid nanomaterial as virus DNA sensing platform. Biosens. Bioelectron. 2018;102:425–431. doi: 10.1016/j.bios.2017.11.052. [PubMed] [CrossRef] [Google Scholar]112. Weerathunge P., Ramanathan R., Torok V., Hodgson K., Xu Y., Goodacre R., Behera B.K., Bansal V. Ultrasensitive colorimetric detection of murine norovirus using NanoZyme aptasensor. Anal. Chem. 2019;91:3270–3276. doi: 10.1021/acs.analchem.8b03300. [PubMed] [CrossRef] [Google Scholar]113. Krammer F., Palese P. Advances in the development of influenza virus vaccines. Nat. Rev. Drug Discov. 2015;14:167–182. doi: 10.1038/nrd4529. [PubMed] [CrossRef] [Google Scholar]114. Moulick A., Richtera L., Milosavljevic V., Cernei N., Haddad Y., Zitka O., Kopel P., Heger Z., Adam V. Advanced nanotechnologies in avian influenza: Current status and future trends—A review. Anal. Chim. Acta. 2017;983:42–53. doi: 10.1016/j.aca.2017.06.045. [PMC free article] [PubMed] [CrossRef] [Google Scholar]115. Sayhi M., Ouerghi O., Belgacem K., Arbi M., Tepeli Y., Ghram A., Anik Ü., Österlund L., Laouini D., Diouani M.F. Electrochemical detection of influenza virus H9N2 based on both immunomagnetic extraction and gold catalysis using an immobilization-free screen printed carbon microelectrode. Biosens. Bioelectron. 2018;107:170–177. doi: 10.1016/j.bios.2018.02.018. [PubMed] [CrossRef] [Google Scholar]116. Tam P.D., Van Hieu N., Chien N.D., Le A.T., Tuan M.A. DNA sensor development based on multi-wall carbon nanotubes for label-free influenza virus (type A) detection. J. Immunol. Methods. 2009;350:118–124. doi: 10.1016/j.jim.2009.08.002. [PubMed] [CrossRef] [Google Scholar]117. Pang Y., Rong Z., Wang J., Xiao R., Wang S. A fluorescent aptasensor for H5N1 influenza virus detection based-on the core–shell nanoparticles metal-enhanced fluorescence (MEF) Biosens. Bioelectron. 2015;66:527–532. doi: 10.1016/j.bios.2014.10.052. [PubMed] [CrossRef] [Google Scholar]118. Vollmer F., Arnold S., Keng D. Single virus detection from the reactive shift of a whispering-gallery mode. Proc. Natl. Acad. Sci. USA. 2008;105:20701–20704. doi: 10.1073/pnas.0808988106. [PMC free article] [PubMed] [CrossRef] [Google Scholar]119. Bai H., Wang R., Hargis B., Lu H., Li Y. A SPR aptasensor for detection of avian influenza virus H5N1. Sensors. 2012;12:12506–12518. doi: 10.3390/s120912506. [PMC free article] [PubMed] [CrossRef] [Google Scholar]120. Mustafa M.S., Rasotgi V., Jain S., Gupta V. Discovery of fifth serotype of dengue virus (DENV-5): A new public health dilemma in dengue control. Med. J. Armed Forces India. 2015;71:67–70. doi: 10.1016/j.mjafi.2014.09.011. [PMC free article] [PubMed] [CrossRef] [Google Scholar]121. Diamond M.S., Pierson T.C. Molecular insight into dengue virus pathogenesis and its implications for disease control. Cell. 2015;162:488–492. doi: 10.1016/j.cell.2015.07.005. [PMC free article] [PubMed] [CrossRef] [Google Scholar]122. Parkash O., Shueb R. Diagnosis of dengue infection using conventional and biosensor based techniques. Viruses. 2015;7:5410–5427. doi: 10.3390/v7102877. [PMC free article] [PubMed] [CrossRef] [Google Scholar]123. Zhang G.J., Zhang L., Huang M.J., Luo Z.H.H., Tay G.K.I., Lim E.J.A., Chen Y. Silicon nanowire biosensor for highly sensitive and rapid detection of Dengue virus. Sens. Actuators B-Chem. 2010;146:138–144. doi: 10.1016/j.snb.2010.02.021. [CrossRef] [Google Scholar]124. Lim J.M., Kim J.H., Ryu M.Y., Cho C.H., Park T.J., Park J.P. An electrochemical peptide sensor for detection of dengue fever biomarker NS1. Anal. Chim. Acta. 2018;1026:109–116. doi: 10.1016/j.aca.2018.04.005. [PubMed] [CrossRef] [Google Scholar]125. Deng J., Toh C.S. Impedimetric DNA biosensor based on a nanoporous alumina membrane for the detection of the specific oligonucleotide sequence of dengue virus. Sensors. 2013;13:7774–7785. doi: 10.3390/s130607774. [PMC free article] [PubMed] [CrossRef] [Google Scholar]126. Jahanshahi P., Zalnezhad E., Sekaran S.D., Adikan F.R.M. Rapid immunoglobulin M-based dengue diagnostic test using surface plasmon resonance biosensor. Sci. Rep. 2014;4:3851–3858. doi: 10.1038/srep03851. [PMC free article] [PubMed] [CrossRef] [Google Scholar]127. Zhang W., Guo S., Carvalho W.S.P., Jiang Y., Serpe M.J. Portable point-of-care diagnostic devices. Anal. Methods. 2016;8:7847–7868. doi: 10.1039/C6AY02158A. [CrossRef] [Google Scholar]128. Heukelbach J., Alencar C.H., Kelvin A.A., de Oliveira W.K., de Goes Cavalcanti L.P. Zika virus outbreak in Brazil. J. Infect. Dev. Ctries. 2016;10:116–120. doi: 10.3855/jidc.8217. [PubMed] [CrossRef] [Google Scholar]129. Inci F., Filippini C., Baday M., Ozen M.O., Calamak S., Durmus S., Wang S. Multitarget, quantitative nanoplasmonic electrical field-enhanced resonating device (NE2RD) for diagnostics. Proc. Natl. Acad. Sci. USA. 2015;112:E4354–E4363. doi: 10.1073/pnas.1510824112. [PMC free article] [PubMed] [CrossRef] [Google Scholar]130. Jin C.E., Lee T.Y., Koo B., Sung H., Kim S.H., Shin Y. Rapid virus diagnostic system using bio-optical sensor and microfluidic sample processing. Sens. Actuators B-Chem. 2018;255:2399–2406. doi: 10.1016/j.snb.2017.08.197. [CrossRef] [Google Scholar]131. Prabowo B.A., Wang R.Y., Secario M.K., Ou P.T., Alom A., Liu J.J., Liu K.C. Rapid detection and quantification of Enterovirus 71 by a portable surface plasmon resonance biosensor. Biosens. Bioelectron. 2017;92:186–191. doi: 10.1016/j.bios.2017.01.043. [PubMed] [CrossRef] [Google Scholar]132. Riedel T., Rodriguez-Emmenegger C., de los Santos Pereira A., Bědajánková A., Jinoch P., Boltovets P.M., Brynda E. Diagnosis of Epstein–Barr virus infection in clinical serum samples by an SPR biosensor assay. Biosens. Bioelectron. 2014;5:278–284. doi: 10.1016/j.bios.2013.12.011. [PubMed] [CrossRef] [Google Scholar]133. Bai W., Spivak D.A. A double-imprinted diffraction-grating sensor based on a virus-responsive super-aptamer hydrogel derived from an impure extract. Angew. Chem. Int. Ed. 2014;53:2095–2098. doi: 10.1002/anie.201309462. [PubMed] [CrossRef] [Google Scholar]134. Inan H., Wang S., Inci F., Baday M., Zangar R., Kesiraju S., Anderson K.S., Cunningham B.T., Demirci U. Isolation, detection, and quantification of cancer biomarkers in HPV-associated malignancies. Sci. Rep. 2017;7:3322–3333. doi: 10.1038/s41598-017-02672-6. [PMC free article] [PubMed] [CrossRef] [Google Scholar]135. Birnbaumer G.M., Lieberzeit P.A., Richter L., Schirhagl R., Milnera M., Dickert F.L., Bailey A., Ertl P. Detection of viruses with molecularly imprinted polymers integrated on a microfluidic biochip using contact-less dielectric microsensors. Lab Chip. 2009;9:3549–3556. doi: 10.1039/b914738a. [PubMed] [CrossRef] [Google Scholar]136. Feng W., Liang C., Gong H., Cai C. Sensitive detection of Japanese encephalitis virus by surface molecularly imprinted technique based on fluorescent method. New J. Chem. 2018;42:3503–3509. doi: 10.1039/C7NJ04791F. [CrossRef] [Google Scholar]
