l’innovazione degli elettrodi di grafene stampati in 3D modificati da AuPs
presso Luiz R. G. Silva 1,2,Jéssica S. Stefano 1,*,Luiz O. Orzari 1,2,Laís C. Brazaca 3,4,Emanuel Carrilho 3,4,Luiz H. Marcolino-Junior 5,Marcio F. Bergamini 5,Rodrigo A. A. Munoz 4,6 eBruno C. Janegitz
*1Dipartimento di Scienze della Natura, Matematica ed Educazione, Università Federale di São Carlos, Araras 13600-970, SP, Brasile
2Dipartimento di Fisica, Chimica e Matematica, Università Federale di São Carlos, Sorocaba 18052-780, SP, Brasile
3Istituto di Chimica di São Carlos (IQSC), Università di San Paolo (USP), São Carlos 13566-590, SP, Brasile
4Istituto Nazionale di Scienza e Tecnologia in Bioanalisi-INCTBio, Campinas 13083-970, SP, Brasile
5Dipartimento di Chimica, Laboratorio di Sensori Elettrochimici (LabSensE), Università Federale del Paraná, Curitiba 81531-980, PR, Brasile
6Istituto di Chimica, Università Federale di Uberlândia, Uberlândia 38400-902, MG, Brasile*Autori a cui indirizzare la corrispondenza.Biosensori2022, 12(8), 622;
https://doi.org/10.3390/bios12080622Ricevuto il: 18 luglio 2022 / Data revisione: 5 agosto 2022 / Accettato: 6 agosto 2022 / Pubblicato: 10 Agosto 2022(Questo articolo appartiene al numero speciale Printed Electrochemical Biosensors)
Astratto
Un elettrodo polilattico di grafene polilattico (G-PLA) a basso costo e usa e getta stampato in 3D modificato con particelle d’oro (AuPs) è stato esplorato per rilevare il cDNA di SARS-CoV-2 e creatinina, un potenziale biomarcatore per COVID-19. Per questo, è stato applicato un semplice sensore elettrochimico non enzimatico, basato su una piattaforma G-PLA modificata da Au. Gli AuP depositati sull’elettrodo sono stati coinvolti in una reazione di complessazione con la creatinina, con conseguente diminuzione della risposta analitica e fornendo così un dispositivo elettroanalitico rapido e semplice. Le caratterizzazioni fisico-chimiche sono state eseguite mediante SEM, EIS, FTIR e voltammetria ciclica. La voltammetria ad onde quadrate è stata impiegata per il rilevamento della creatinina e il sensore ha presentato una risposta lineare con un limite di rilevamento di 0,016 mmol L−1. Infine, è stato sviluppato un biosensore per il rilevamento di SARS-CoV-2 basato sull’immobilizzazione di una sequenza di cattura del cDNA virale sull’elettrodo stampato in 3D modificato dall’Au. La concentrazione, il tempo di immobilizzazione e il tempo di ibridazione sono stati valutati in presenza del bersaglio del DNA, risultando in un biosensore con analisi rapida e a basso costo, in grado di rilevare il cDNA del virus con un buon limite di rilevazione (0,30 μmol L−1), e alta sensibilità (0,583 μA μmol−1 L). Sono stati ottenuti risultati riproducibili (RSD = 1,14%, n = 3), attestando le potenzialità delle piattaforme stampate in 3D per la produzione di biosensori.Parole chiavi: elettrochimico (bio)sensore; Elettrodo stampato in 3D; Elettrodo modificato AuP; SARS-CoV-2; creatinina
1. Introduzione
Versatilità, libertà di progettazione e basso costo sono caratteristiche differenziali della costruzione di sistemi e dispositivi analitici [1]. In tale contesto, l’uso della tecnologia di stampa 3D è molto attraente grazie alla sua capacità di convertire i processi di produzione convenzionali e centralizzati in un processo di prototipazione rapido, in laboratorio e personalizzabile, consentendo l’ottenimento di un’ampia varietà di strutture in modo semplice [2]. L’uso della modellazione a deposizione fusa (FDM) nelle stampanti 3D fornisce oggetti tridimensionali dopo la deposizione di filamenti termoplastici strato per strato, attraverso un ugello riscaldato [3]. L’emergere di filamenti conduttivi ha permesso l’uso della stampa 3D per la preparazione di dispositivi elettrochimici. Questi filamenti sono solitamente composti da nerofumo (CB) o grafene (G) come materiali conduttivi e polimeri come l’acido polilattico (PLA) [4]. La produzione di sensori stampati in 3D basati sul grafene è stata ampiamente esplorata ed è un’opzione interessante. Le proprietà fisiche ed elettrochimiche uniche di questo materiale forniscono caratteristiche adeguate per essere utilizzate come sensori elettrochimici. Tra queste proprietà, si possono menzionare le caratteristiche meccaniche, l’aumento della superficie e l’eccellente attività elettrochimica con il potenziale per produrre dispositivi altamente sensibili [5,6,7]. Una volta commercializzati questi filamenti, i sensori elettrochimici possono essere facilmente ottenuti. Questi materiali sono presentati come promettenti per lo sviluppo di diversi (bio)sensori, anche se è presente un’elevata quantità di materiale isolante, che richiede fasi di pre-trattamento prima del loro utilizzo [3,8].Sebbene l’uso di elettrodi stampati in 3D per applicazioni di rilevamento sia relativamente nuovo e sia ancora in fase di miglioramento dall’esplorazione di diversi materiali, il potenziale di questi dispositivi come piattaforme per la costruzione di biosensori è già stato dimostrato, fornendo risultati interessanti [9]. Ad esempio, Muñoz e Pumera (2021) hanno riportato la produzione di immunosensori stampati in 3D a base di grafene / PLA modificati con nanoparticelle d’oro per il rilevamento della proteina spike del virus SARS-CoV-2 [10]. Nello stesso anno, Martins e colleghi hanno sviluppato un immunosensore stampato in 3D basato su CB / PLA per determinare la nucleoproteina hantavirus Araucaria [10]. Pertanto, l’uso di elettrodi stampati in 3D come piattaforma per il biorilevamento delle malattie è promettente e ha guadagnato spazio per la crescita e lo sviluppo di una nuova linea di ricerca.Negli ultimi decenni, le malattie infettive trasmesse dai virus sono diventate una preoccupazione significativa in tutto il mondo [11]. L’emergere di nuovi virus, così come la loro mutevolezza, è comune. Nel 2019, il mondo intero è rimasto sorpreso dall’inizio di quella che sarebbe diventata una nuova pandemia globale, causata da un virus respiratorio. Questa sindrome respiratoria acuta grave coronavirus 2 (SARS-CoV-2), responsabile della causa della malattia da coronavirus 2019 (COVID-19), è stata segnalata per la prima volta a Wuhan, in Cina [12].Poiché uno dei modi più efficaci per controllare la diffusione di SARS-CoV-2 si basa sull’isolamento dei pazienti infetti, è necessario sviluppare metodi di rilevamento della malattia rapidi, affidabili e semplici in grado di soddisfare la domanda e identificare individui asintomatici [13]. Diversi lavori hanno riportato lo sviluppo di dispositivi point-of-care per la diagnosi di COVID-19 [14]. Tuttavia, il metodo di diagnosi standard per SARS-CoV-2 si basa sull’uso della reazione a catena della polimerasi (PCR) per amplificare il materiale genetico per il rilevamento del virus [15]. La PCR è altamente sensibile e affidabile, ma presenta dei limiti. Il tempo medio per la generazione dei risultati è basso (24-72 ore) e il tempo tra la raccolta e la consegna dei campioni è imprevedibile a causa della portata della pandemia globale di COVID-19 [16]. Inoltre, questi metodi richiedono professionisti ben addestrati, laboratori ben attrezzati e strumenti sofisticati. In questo contesto, l’implementazione di un metodo di rilevamento in tempo reale a basso costo per lo screening di SARS-CoV-2 è di alta priorità.Sotto questo aspetto, l’uso di metodi elettrochimici per la rilevazione di questo virus è un’alternativa interessante [17]. Sono già stati segnalati diversi tipi di biosensori elettrochimici per il rilevamento di SARS-CoV-2 e altri virus, come aptasensori, immunosensori e sensori di DNA [18,19,20,21,22,23]. Tra questi, si può menzionare il lavoro di Li et al. [22], 2015, che ha sviluppato un sensore di DNA elettrochimico cartaceo per il rilevamento del virus dell’epatite B. Ghanbari et al. [21] 2017 hanno sviluppato un nuovo aptasensor elettrochimico per il rilevamento ultrasensibile del virus dell’epatite C. Per quanto riguarda il rilevamento di SARS-CoV-2, Brazaca et al. [23], nel 2022, hanno sviluppato un immunosensore a basso costo (< US $ 0,03 per dispositivo) basato su elettrodi di carbonio serigrafati modificati in oro per il rilevamento della proteina spike (S) del virus SARS-CoV-2. Tuttavia, solo pochi esplorano l’uso della tecnologia di stampa 3D. Recentemente, il nostro gruppo di ricerca ha affrontato l’ottenimento di nuovi filamenti conduttivi per la stampa 3D di sensori elettrochimici, composti da grafite / PLA (40% p / p) come piattaforma per lo sviluppo di un nuovo immunosensore per la rilevazione di SARS-CoV-2 [24]. L’immunosensore è stato fabbricato attraverso l’immobilizzazione degli anticorpi della proteina spike SARS-CoV-2 direttamente sull’elettrodo non modificato e, poiché la superficie ottenuta presentava i gruppi funzionali, è stata eliminata la necessità di modificare la superficie con particelle metalliche. Il biosensore è stato in grado di rilevare la proteina spike (S1) del virus con elevata sensibilità (0,01 μA μg−1 mL), consentendo il rilevamento della proteina spike (S1) del virus in campioni di saliva sintetica. D’altra parte, come presentato in questo lavoro, l’uso di Au può migliorare la conduttività elettrica e aumentare la superficie, promuovendo caratteristiche interessanti per l’applicabilità degli elettrodi nel biorilevamento.Lo sviluppo di genosensori è apprezzabile dal punto di vista clinico e dal punto di vista della salute collettiva, consentendo il monitoraggio e la diagnosi delle malattie infettive. Questi sensori si basano su modifiche superficiali con materiali genetici specifici che causano cambiamenti nelle proprietà misurabili dopo l’ibridazione [25]. In tal modo, utilizzando i genosensori, il materiale genetico del virus (RNA o cDNA) può essere rilevato utilizzando tecniche elettrochimiche per determinare il tasso di ibridazione di specifici filamenti di cDNA con le loro sonde complementari immobilizzate sulla superficie [20,26,27]. Per quanto ne sappiamo, i lavori in letteratura che riportano lo sviluppo di genosensori stampati in 3D per il rilevamento di SARS-CoV-2 sono scarsi fino ad oggi. Pertanto, l’uso di piattaforme stampate in 3D per il rilevamento di COVID-19 deve ancora essere esplorato. Nel lavoro sviluppato da Crevillen et al. [28] è stato riportato l’uso di elettrodi 3D stampati a penna (3D-PP), composti da grafene / PLA per lo sviluppo di un genosensore mediante adsorbimento della sonda (ssDNA), che ha preso di mira la sequenza genica N del virus SARS-CoV-2. L’ibridazione di ssDNA e RNA virale ha causato il desorbimento di ssDNA sulla superficie dell’elettrodo e si è verificata l’ossidazione elettrochimica dell’adenina presente nella sonda non desorbita, fornendo una risposta voltammetrica. Il genosensore è stato integrato in un canale microfluidico PDMS, fornendo un sistema lab-on-a-chip, in grado di rilevare l’RNA di SARS-CoV-2.Inoltre, il rilevamento di biomarcatori per SARS-CoV-2 può essere un approccio interessante per migliorare la diagnosi clinica. COVID-19 è stato comunemente associato a danni renali, che possono essere diagnosticati da livelli elevati di creatinina (CNN) [29,30]. La CNN è prodotta nei muscoli dalla creatina, rilasciata nel flusso sanguigno ed escreta dal rene [31]. La misurazione dei livelli di CNN nel sangue umano o nelle urine è clinicamente essenziale perché i livelli riflettono parzialmente lo stato della funzione renale e muscolare e alti livelli di creatinina (superiori a 150,0 o 500,0 μmol L−1) indicano un malfunzionamento dei reni [31,32,33]. Valori inferiori a 40,0 μmol L−1 sono indicativi di diminuzione della massa muscolare [33,34,35,36]. È anche estremamente importante monitorare la presenza di questo possibile biomarcatore COVID-19 poiché può indicare un’infezione precoce del paziente da parte del virus [30,37].In questo scenario, i sensori elettrochimici sono presentati come dispositivi analitici interessanti perché combinano alta sensibilità, semplicità di funzionamento e basso costo [38]. In letteratura, vari tipi di sensori enzimatici [39] e non enzimatici [40,41] sono riportati per la determinazione della CNN con diverse tecniche elettrochimiche [42,43,44]. Sebbene selettivi, i biosensori enzimatici presentano alcune limitazioni rispetto agli elettrodi non enzimatici. In questo contesto, l’alto costo e la denaturazione, la preparazione dispendiosa in termini di tempo e la mancanza di stabilità possono essere evidenziati come punti negativi che possono essere sagomati quando vengono applicati sensori non enzimatici. Pertanto, lo sviluppo di sensori non enzimatici per il rilevamento della CNN è un’opzione altamente praticabile e vantaggiosa [40,45,46]. Tuttavia, mancano ancora opere che esplorano l’uso di dispositivi miniaturizzati e portatili per la determinazione della CNN e l’uso di dispositivi stampati in 3D per il rilevamento della CNN non è stato riportato in letteratura fino ad ora.In questo aspetto, la versatilità nel processo produttivo che impiega la tecnica di stampa 3D [3,47,48,49], legata all’uso di metalli elettrocatalitici elettrodepositati sulla superficie dell’elettrodo, può migliorare la sensibilità, la stabilità e l’applicabilità nei dispositivi elettroanalitici. Si desidera l’uso di particelle d’oro per la modifica degli elettrodi, per la sua buona conduttività, biocompatibilità e proprietà chimiche ed elettrocatalitiche, e per essere materiali interessanti per la modifica di sensori elettrochimici (bio)[50,51,52].Pertanto, in questo lavoro, presentiamo per la prima volta lo sviluppo di un biosensore di cDNA stampato in 3D per il rilevamento di SARS-CoV-2 nel siero umano e nella saliva sintetica utilizzando una piattaforma stampata in 3D semplice, rapida, a basso costo, miniaturizzata e versatile a base di grafene / PLA modificata con particelle Au. Inoltre, abbiamo esplorato la piattaforma proposta per il rilevamento del segnale di spegnimento del biomarcatore CNN nelle urine sintetiche e nel siero umano.
2. Materiali e metodi
L’acqua ultrapura di un sistema Milli-Q Plus (Millipore Corporation, Darmstadt, Germania) è stata utilizzata per preparare tutte le soluzioni acquose. Il cloruro di sodio (NaCl) (98%, Vetec, Rio de Janeiro, Brasile) è stato utilizzato come elettrolita di supporto nelle analisi voltammetriche per determinare la CNN. La modifica dell’elettrodo è stata eseguita con cloruro d’oro (III) triidrato (99%, Sigma-Aldrich, St. Louis, MO, USA) in acido solforico (H®®®2Così4) (95%, Vetec, Brasile). I test di interferenza sono stati eseguiti utilizzando glucosio, acido ascorbico (AA) (Sigma-Aldrich, USA), acido urico (AU) (99%, Sigma-Aldrich, USA) e glutatione ridotto (98%, Sigma-Aldrich, USA). A 10,0 mmol L®®®®−1 la soluzione madre di creatinina è stata utilizzata come standard analitico.Entrambi i sintetici (urina e saliva) sono stati preparati seguendo i protocolli della letteratura. L’urina sintetica è stata prodotta con NaCl (98%, Vetec, Brasile), cloruro di potassio (99%, Vetec, Brasile), cloruro di calcio diidrato (99%, Vetec, Brasile), solfato di sodio anidro (99%, Vetec, Brasile), fosfato monopotassico (99%, Vetec, Brasile), cloruro di ammonio (99%, Vetec, Brasile) e urea (99%, Dinamica, Indaiatuba, Brasile) [53]. La saliva sintetica è stata preparata con cloruro di sodio (99%, Vetec, Brasile), fosfato di sodio bibasico (99%, Vetec, Brasile), cloruro di potassio (≥99%, Vetec, Brasile), tiocianato di potassio (99%, Vetec, Brasile) e urea [54]. Il siero umano è stato acquistato da Sigma-Aldrich, USA.®®®®®®®®®®®®Per lo sviluppo del biosensore e ulteriori analisi, sono stati acquisiti 2-mercaptoetanolo e Tris Buffered Saline 10× (Sigma-Aldrich, USA). Sono state ottenute tre sequenze di DNA (EXXTEND, Paulinia, Brasile): la cattura della sequenza, o sonda (Thiol C6-AGATGTCTTGTGCTGCCGGTA), ottenuta dal gene ORF1ab, il bersaglio della sequenza (TAGCCGGCAGCACAAGACATCT) e il target della sequenza di controllo negativo (TGACTACAGAAGTGGCTTTTG).®®
2.1. Apparecchi, celle elettrochimiche ed elettrodi
Tutte le misurazioni elettrochimiche sono state eseguite utilizzando un potenziostato/galvanostato PGSTAT204 Metrohm (Eco Chemie, Utrecht, Paesi Bassi) guidato dal software NOVA (versione 2.1.4), utilizzato anche per l’acquisizione e il trattamento dei dati. La correzione dello sfondo è stata utilizzata per il rilevamento voltammetrico della CNN per una migliore risoluzione di picco, utilizzando l’algoritmo “media mobile”, con la dimensione della finestra impostata su 2.®Una stampante 3D Sethi3D S3 (Campinas, Brasile), alimentata sia da filamenti termoplastici conduttivi pla (drogati con grafene) acquisiti da Black Magic 3D (New York, NY, USA) che non conduttivi (PLA da Sethi3D (Campinas, Brasile)), sono stati utilizzati per la produzione del sistema elettrochimico completamente stampato in 3D, con il metodo FDM.®®
2.2. Caratterizzazione morfologica ed elettrochimica
Microscopia elettronica a scansione (SEM) Thermo Fisher Modello scientifico Prisma E con tecnologia ColorSEM e spettroscopia a raggi X a dispersione di energia integrata (EDX) è stato utilizzato per acquisire immagini SEM. Le immagini sono state ottenute per G-PLA e Au/G-PLA. Spettroscopia infrarossa e risultati sono stati ottenuti da uno spettrometro a infrarossi a trasformata di Fourier Bruker ALPHA II tra 4000 e 600 cm®−1.Sono state eseguite analisi di spettroscopia di impedenza elettrochimica (EIS) per comprendere i diversi effetti superficiali provocati dall’aggiunta di Au su elettrodi di lavoro al grafene. Ogni studio è stato condotto applicando i suoi specifici valori di potenziale a circuito aperto (0,6 e 9,0 mV per elettrodi G-PLA e Au/G-PLA, rispettivamente), misurati dopo una stabilizzazione di 300 s, con un raccordo χ2 di 0,04367 (G-PLA) e 0,0272 (Au/G-PLA).
2.3. Produzione dell’elettrodo G-PLA
Gli elettrodi di riferimento, di lavoro e di controelettrodi sono stati fabbricati con una stampante 3D, con il design basato su SPE commerciali (elettrodi serigrafati), in cui l’elettrodo di lavoro consisteva in un cerchio di 4 mm di diametro, il controelettrodo consisteva in un semicerchio di 9 mm, che era posto intorno all’elettrodo funzionante, e l’elettrodo di riferimento consisteva in un semicerchio più piccolo per riempire lo spazio intorno all’elettrodo di lavoro, come mostrato nello schema S1. Tutti e tre gli elettrodi sono stati fabbricati sulla punta di un rettangolo di 1,5 cm, responsabile del collegamento con i cavi potenziostatici, che sono stati isolati con smalto per unghie. Gli elettrodi sono stati stampati in 3D utilizzando un filamento G-PLA commerciale e i disegni sono stati disegnati utilizzando Blender e, infine, importati dal software Simplify3D, che controlla la stampante 3D. I file STL ottenuti sono disponibili sul sito web della rivista. Infine, gli elettrodi sono stati stampati a 190 °C. Dopo la stampa, gli elettrodi sono stati assemblati secondo lo schema S1. Inizialmente, gli elettrodi sono stati fissati sul supporto (foglio di plastica dura) e sopra il nastro biadesivo, che è stato delimitato con smalto incolore (circa la metà del contatto elettrico) e asciugato per 20 minuti.
2.4. Ottimizzazione ed elettrodeposizione di Au
L’ottimizzazione dell’elettrodeposizione di Au sul G-PLA è stata condotta con un 22 progettazione composita centrale (CCD) con tre repliche sul punto centrale. L’elettrodeposizione di Au è stata eseguita applicando costantemente -0,6 V. Le risposte utilizzate nell’ottimizzazione sono state ottenute dalla CNN (3,0 mmol L−1) in NaCl (0,5 mol L−1) analisi tramite SWV con i parametri di funzionamento standard del software (potenziale di passo (5 mV); ampiezza di modulazione (20 mV) e frequenza (15 Hz)). In questo passaggio, le variabili di interesse (tempo di deposizione (X1) e concentrazione di Au (X2)) were studied in a range of 47 to 683 s, and 0.76 to 9.25 mmol L−1. The obtained response surface was then used for the establishment of the best electrodeposition conditions. The experimental layout of the 22 CCD with the experiments performed, variables and their respective actual and normalized levels, and CNN analytical signal response can be found in Table S1 (Supplementary Material). Therefore, the SWV parameters were set at 5 mV (step potential), 20 mV (modulation amplitude), and 10 Hz (frequency).
2.5. Optimization of SWV Variables for CNN
The pre-optimization of the operational parameters (step potential; modulation amplitude; and frequency) of the SWV technique was performed with a 23 full factorial design at a range of 2.0 to 10 mV, 20 to 60 mV, and 6.0 to 34 Hz, respectively. This procedure evaluated the significance of the three SWV variables in a solution containing 3.0 mmol L−1 CNN and 0.5 mol L−1 NaCl. Table S2 shows the 23 full factorial designs (variables and their respective actual and normalized levels, and CNN analytical signal response). The experiments in Table S2 were performed in a random order [55,56].Subsequently, the two significant variables (step potential and frequency) were optimized by applying a 22 CCD at a range of (1.0 to 7.0 mV and 6.0 to 34 Hz) in a solution of 5.0 mmol L−1 CNN in 0.5 mol L−1 NaCl. The 22 CCD matrix of the optimization step can be found in Table S3.
2.6. Biosensor Preparation
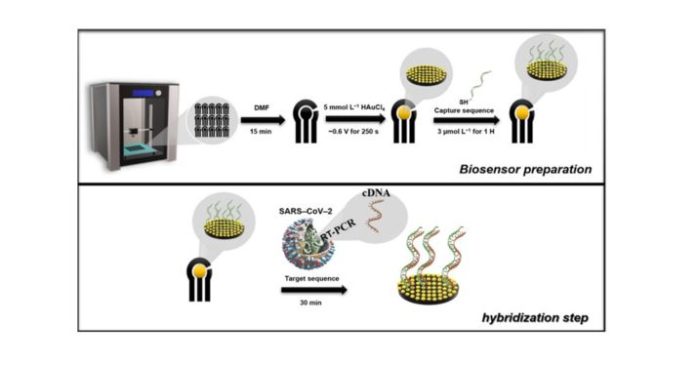
The biosensor was prepared using the G-PLA sensor previously modified with Au by electrodeposition. For the development of the biosensor, the Au deposition time was also optimized, and the optimization was performed using univariate experiments applying −0.6 V constantly, in which the concentration was fixed at 5.0 mmol L−1, after the previous optimization. The deposition time varied from 100 to 300 s and the cyclic voltammetry technique was selected for the analysis employing a 1.0 mmol L−1 ferrocenemethanol redox probe in 0.1 mol L−1 KCl.Subsequently, the Au/G-PLA was the base for the immobilization of the cDNA capture sequence by the drop-casting method, in which the immobilization time and concentration were optimized. The capture cDNA immobilization occurs because the thiol groups present on the cDNA strand bind to the gold particles present on the surface of the 3D-printed sensor, allowing the capture strand to be anchored. Initially, a solution containing 100.0 µmol L−1 of the capture sequence and 0.12 mmol L−1 2-mercaptoethanol in 10.0 mmol L−1 TRIS buffer was prepared as a stock solution. All subsequent dilutions were carried out with ultra-pure water following a previous protocol from the literature [17]. The optimization method was multivariate, using the central composite design, employing a range from 1.0 to 7.0 µmol L−1 of capture sequence (X1) and hours (X2) for both parameters. The variables, their respective actual and normalized levels, and their analytical signal responses (current difference between the Au/G-PLA signal to the biosensor employing 1.0 mmol L−1 ferrocenemethanol in 0.1 mol L−1 KCl) can be found in Table S4 (Supplementary Material).For analysis of the target sequence, a drop containing the biomolecule was cast on the surface of the biosensor. It is expected that the target sequence hybridizes with the capture sequence immobilized on the surface of the electrode. The hybridization time was optimized by univariate experiments, varying the time from 30 to 180 min with a concentration of 1.0 µmol L−1 of the target sequence. The biosensor production, as well as the hybridization step (determination of the target sequence), was present in Scheme 1, and for better comprehension of the whole process, a time-lapsed video showing the 3D printing of the electrodes and biosensor production steps and the STL files for 3D printing are available on the journal website.
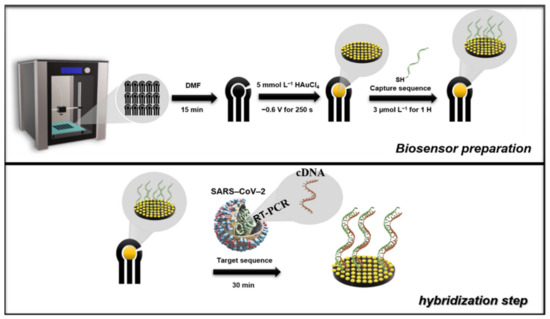
Scheme 1. Schematic illustration of the production of the biosensor and hybridization step. The production of the biosensor consists of the printing step, chemical treatment of the surface, modification with Au (5.0 mmol L−1), and, finally, modification of the surface with the capture sequence (3.0 µmol L−1 for 1 h). The hybridization step is carried out for 30 min after adding the drop of solution containing the target sequence to the surface of the biosensor.
3. Results and Discussion
3.1. Caratterizzazione morfologica ed elettrochimica
Le morfologie degli elettrodi G-PLA e Au/G-PLA (deposizione 250 e 400 s) sono state caratterizzate da SEM. Gli spettri FT-IR di G-PLA e Au/G-PLA sono stati registrati nell’intervallo di numero d’onda da 600 a 4000 cm−1. La Figura 1 presenta le immagini SEM ottenute per gli elettrodi proposti e gli spettri FT-IR.
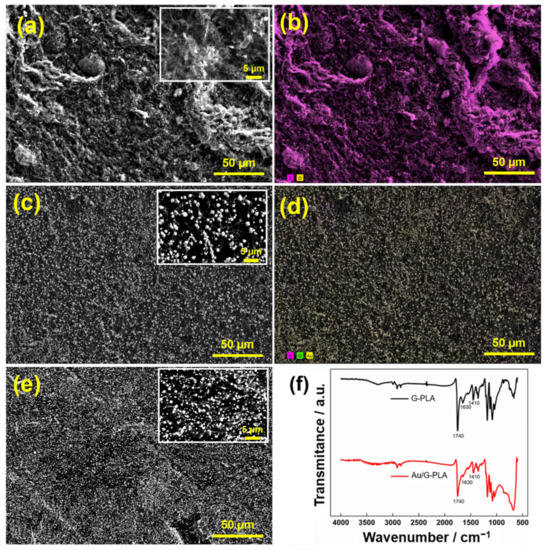
Figura 1. Le immagini SEM delle superfici degli elettrodi (a) G-PLA, (b) la mappatura degli elementi corrispondenti di G-PLA, (c) Au/G-PLA (deposizione di 400 s), (d) la mappatura degli elementi corrispondenti di Au/G-PLA, (e) Au/G-PLA (deposizione di 250 s) e (f) gli spettri FT-IR di G-PLA (nero) e Au/G-PLA (rosso) sono stati registrati entro l’intervallo di numero d’onda compreso tra 600 e 4000 cm−1.La Figura 1a presenta la morfologia superficiale ottenuta dall’analisi SEM dell’elettrodo G-PLA. Si può osservare una superficie non uniforme, con grandi irregolarità superficiali, probabilmente causate dall’esposizione di materiale grafene dopo il trattamento superficiale. Le immagini superficiali di Au/G-PLA mostrano, come previsto, particelle d’oro (AuP) ben distribuite su tutta la superficie del sensore. Inoltre, diversi tempi di deposizione hanno mostrato una diversa distribuzione e numero di AUP sulla superficie. Pertanto, l’ottimizzazione di questo parametro era necessaria per ogni tipo di applicazione desiderata. Inoltre, la mappatura degli elementi in Figura 1b mostra la presenza di carbonio sulla superficie e la Figura 1d mostra che la presenza di Au è predominante. D’altra parte, gli spettri FTIR del G-PLA presentano i picchi caratteristici a 1410, 1630 e 1740 cm−1, che può essere associato a C-H2 legami, e legami C=C (anello aromatico) e legami C=O (carbossil/carbonile), rispettivamente [57]. Queste bande sono osservate a causa della presenza di PLA sulla superficie dell’elettrodo. Dopo la deposizione di Au sulla superficie del G-PLA, i picchi si riferivano a C=O, C=C e C-H2 legami, ha presentato un cambiamento nell’intensità (diminuzione), indicando il coordinamento tra i legami e Au. Pertanto, l’FTIR conferma che l’elettrodeposizione di Au si è verificata con successo sulla superficie del G-PLA [58]. Inoltre, lo spettro EDX è mostrato nella Figura S1, che dimostra la presenza di carbonio, ossigeno e Au sulla superficie del sensore, con percentuali atomiche di 64,8, 32,1 e 3,1, rispettivamente.La Figura S2 mostra i grafici di Bode sia per gli elettrodi che per il diagramma di Nyquist di questi sistemi. L’inserto presenta tutte le informazioni di interesse, compresi i circuiti Randles modificati equivalenti. Nella Figura S2a,b, troviamo i grafici di Bode per entrambi gli elettrodi. Come si può notare a frequenze più elevate, c’è un leggero cambiamento nella resistenza elettrolitica (Rs, da 165 a 144 Ω). Tuttavia, considerando la differenza di potenziale applicata, i risultati hanno suggerito che l’elettrodeposizione di Au non ha alcun effetto di carica significativo nella formazione del doppio strato. Entrambi i sistemi presentavano massime a valori di fase inferiori a π/2°, il che indica un’impedenza controllata principalmente dal fattore di resistenza, soprattutto dopo l’elettrodeposizione di Au. Nella Figura S2c, il sistema Au/G-PLA mostra una resistenza al trasferimento di carica notevolmente inferiore (RCt) rispetto all’elettrodo nudo. Il processo capacitivo minore suggerisce che il primo potrebbe essere più sensibile alle reazioni elettrochimiche rispetto all’elettrodo di grafene nudo.Infine, è stata calcolata la superficie attiva degli elettrodi per l’elettrodo G-PLA e il G-PLA modificato da Au. Per questo, registrazioni voltammetriche cicliche a diverse velocità di scansione (20, 40, 60, 80, 100, 120, 140, 160, 200 e 250 mV s−1) sono stati eseguiti su entrambi gli elettrodi, utilizzando un L 1,0 mmol−1 soluzione di ferrocenmetanolo in 0,1 mol L−1 Kcl. L’area elettroattiva è stata calcolata usando l’equazione di Randles-Ševčík:Iop = 2,69 × 105A C D1/2n3/2v1/2dove Ip è la corrente di picco (A), A l’area elettroattiva (cm2), C la concentrazione della sonda redox (mol L−1), D il coefficiente di diffusione della sonda redox (cm2 s−1), n il numero di elettroni coinvolti nella reazione e v la velocità di scansione (mV s−1). La figura S3 mostra i voltammogrammi ottenuti per G-PLA e Au/G-PLA con il rispettivo grafico della risposta corrente in funzione di v1/2. I valori ottenuti per G-PLA e Au/G-PLA sono stati 0,16 e 0,22 cm2, rispettivamente, indicando che la deposizione di Au fornisce un aumento dell’area attiva. Questo risultato è in accordo con la riduzione della RCt valori osservati negli studi EIS.
3.2.3D sensore Au/G-PLA stampato per il rilevamento della CNN
3.2.1. Voltammetria ciclica
La risposta elettrochimica dell’Au/G-PLA è stata valutata utilizzando la voltammetria ciclica (CV) in presenza e assenza di 2,0 mmol L−1 CNN in 0,5 mol L−1 NaCl. La Figura 2 mostra i voltammogrammi ciclici ottenuti.
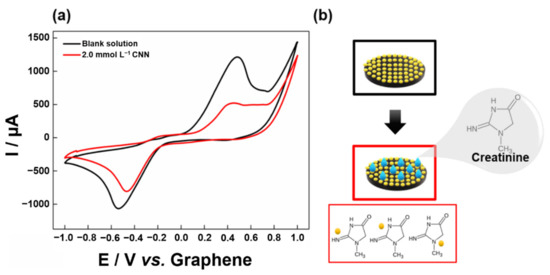
Figura 2.a) Voltammogrammi ciclici di soluzione bianca (linea nera) e (linea rossa) 2,0 mmol L−1 CNN utilizzando elettrodo Au/G-PLA. Elettrolita di supporto: 0,5 mol L−1 NaCl. Velocità di scansione: 100 mV s−1, (b) Illustrazione schematica dell’interazione della creatinina con AuPs.Nella Figura 2a, è possibile osservare che l’elettrodo Au/G-PLA presentava un tipico profilo di processo redox in voltammetria ciclica, con picchi catodici (−0,5 V) e anodici (0,4 V) corrispondenti all’Au presente sulla superficie dell’elettrodo. In presenza di 2,0 mmol L−1 CNN, il segnale di corrente Au decade in modo significativo, dimostrando che l’interazione tra l’Au presente sulla superficie del sensore e la CNN avviene con successo (Figura 2b). Secondo la letteratura, la CNN contiene tre gruppi di azoto nella sua struttura e l’Au presente sulla superficie dell’elettrodo può legarsi a composti di azoto ricchi di elettroni attraverso l’interazione tra gli atomi N e Au [59,60]. Inoltre, è interessante osservare che le ammine primarie sono generalmente utilizzate per modificare la superficie degli elettrodi Au (principalmente AuPs), e l’anello di azoto degli aromatici ibridi mostra una più forte affinità di legame per i metalli Au [61,62,63]. Pertanto, la determinazione elettrochimica della CNN è possibile utilizzando l’elettrodo proposto.
3.2.2. Ottimizzazione ed elettrodeposizione di Au
Pertanto, è stata eseguita un’ottimizzazione dell’elettrodeposizione di Au sul sensore G-PLA stampato in 3D in base alla più alta risposta di corrente per cnn (differenza tra segnale Au / G-PLA in assenza e presenza di CNN) e sono stati studiati i parametri di concentrazione e tempo di deposizione. Dalle risposte presentate nella matrice CCD (Tabella S1), è stato possibile costruire la superficie di risposta e la curva di livello (Figura 3a,b) per ottenere le migliori condizioni per l’elettrodeposizione, raggiungendo l’Au/G-PLA ottimizzato per la determinazione della CNN.
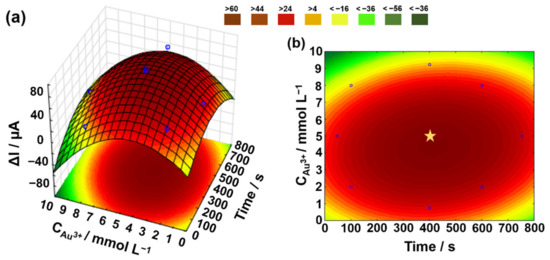
Figura 3. (a) Risposta superficiale e (b) curva di livello ottenuta per l’ottimizzazione delle variabili: Au3+ concentrazione (mmol L−1) e tempo (i) di elettrodeposizione in funzione della corrente in presenza di 3,0 mmol L−1 Cnn.Analizzando la Figura 3, è stata identificata la regione di corrente “massima” (segnata dalla stella in Figura 3b), mostrando una regione stabilizzata con un intervallo di lavoro ottimale per le variabili coinvolte. I valori ottimali scelti per lo sviluppo dell’Au/G-PLA per la determinazione della CNN sono stati 5,0 mmol L−1 e 400 s per la concentrazione di Au e il tempo di elettrodeposizione, rispettivamente.
3.2.3. Ottimizzazione delle variabili SWV
Sulla base delle condizioni ottimizzate per l’elettrodeposizione Au, lo screening delle variabili SWV (potenziale di passo, ampiezza di modulazione e frequenza) è stato eseguito mediante un progetto fattoriale completo (23). Gli effetti calcolati (potenziale di passo (X1), ampiezza di modulazione (X2), e frequenza (X3)) delle risposte ottenute nella tabella S2 è visibile nella tabella S5.L’effetto calcolato della variabile di ampiezza di modulazione (X2) è stato considerato insignificante, anche se non è passato attraverso il valore “zero” nell’intervallo di confidenza. Tuttavia, l’effetto calcolato di questo parametro presentava un valore vicino al valore dell’effetto del terzo ordine calcolato (X123), che a sua volta è considerato trascurabile [55,56,64]. Pertanto, la variabile di ampiezza di modulazione è stata impostata su un valore di 20 mV (punto centrale) e le altre due variabili sono state selezionate e studiate in CCD.Utilizzando le risposte ottenute e presentate nella Tabella S3, generiamo il modello e la costruzione della superficie di risposta e della curva di livello, illustrando il comportamento della corrente di picco in presenza di CNN per quanto riguarda la frequenza × il potenziale di passo. La Figura S4 mostra la superficie di risposta e la curva di livello ottenuta. Analizzando la Figura S4, è stata identificata la regione di sovrapposizione per il massimo ΔI della CNN. Pertanto, i valori ottimali selezionati sono 5,0 mV e 10 Hz rispettivamente per potenziale di passo e frequenza.
3.2.4. Curva analitica
Dalle condizioni precedentemente ottimizzate del SWV, è stata costruita una curva analitica per CNN in un intervallo di concentrazione compreso tra 0,050 e 3,2 mmol L−1 (Figura 4).
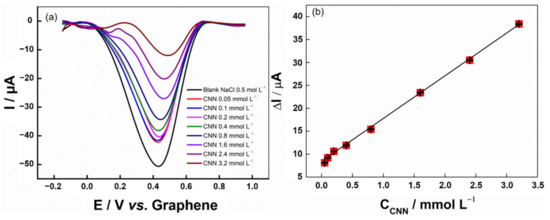
Figura 4.(a) Voltammogrammi ad onda quadra per l’aggiunta di otto concentrazioni di CNN (da 0,05 a 3,2 mmol L−1) in 0,5 mol L−1 NaCl. Parametri SWV: −5,0 mV (potenziale di passo); 20 mV (ampiezza di modulazione); 10 Hz (frequenza), (b) La curva analitica è stata ottenuta dalla variazione di Icima in funzione della concentrazione della CNN.Dalla curva generata, il coefficiente di determinazione (R2) ottenuto è stato 0,998 nell’intervallo di concentrazione da 0,05 a 3,2 mmol L−1, che ha fornito l’equazione ΔI (μA) = 8,46 + 9,318 × CCnn (mmol L−1). I valori di precisione intra-elettrodo (n = 3) e intra-giorno (n = 3) (%RSD) erano rispettivamente del 4,1% e del 3,7%, ottenuti per misurazioni utilizzando una concentrazione di 0,1 mmol L−1 Cnn. I limiti di rilevazione (LOD) e di quantificazione (LOQ) sono stati calcolati secondo le formule LOD = (3,3 × SDintercettare)/b e LOQ = 3 × LOD, dove SDintercpt è la deviazione standard di tre curve analitiche realizzate con l’intervallo lineare presentato e b è la sensibilità della curva di calibrazione ottenuta dal valore medio dei triplicati. I valori loq e LOD sono stati stimati in 0,05 e 0,02 mmol L−1rispettivamente. Il LOD trovato per la procedura proposta può determinare la CNN a livelli abbastanza bassi da indicare problemi di salute (>150 μmol L−1), come una grave compromissione renale, che alla fine porta alla dialisi o al trapianto [33,34,35,36].La tabella S6 presenta un confronto tra i dati ottenuti dall’elettrodo stampato in 3D proposto e altri sensori basati sulle piattaforme non enzimatiche ed enzimatiche (biosensori) per il rilevamento della CNN. Si può osservare che i lavori confrontati presentano intervalli lineari simili e LOD. Tuttavia, alcuni dei lavori pubblicati richiedono l’associazione di enzimi per il rilevamento della CNN o modifiche più complesse, rendendoli più laboriosi e più costosi dell’elettrodo stampato in 3D proposto. Le buone prestazioni analitiche dell’elettrodo stampato in 3D rendono la sua applicazione attraente, poiché la produzione su larga scala è possibile, a un costo relativamente basso. L’elettrodo proposto è semplice da preparare e fornisce un’analisi rapida.
3.2.5. Test di interferenza e recupero
È stato inoltre effettuato uno studio di interferenza per valutare l’interferenza causata da altre specie nel segnale SWV di 0,7 mmol L−1 Cnn. Lo studio è stato eseguito registrando SLV in presenza di glucosio, AA, AU e glutatione ridotto, nel rapporto di 1:5 (CNN: interferente). La tabella S7 mostra la risposta analitica ottenuta per questo studio. È possibile osservare che l’attuale risposta della CNN ottenuta in presenza degli interferenti variava dal 94,70 al 109,9%. Pertanto, non sono stati osservati cambiamenti significativi nel segnale analitico della CNN. Inoltre, i risultati sono in accordo con quelli presentati da Fava et al. 2020 [40].Un test di recupero analitico ha verificato la precisione della procedura utilizzando SWV associato ad Au/G-PLA e la possibilità di interferenze dalla matrice (urina sintetica e siero umano). Per questa fase, i campioni di urina sintetica e siero umano sono stati arricchiti con tre diverse concentrazioni di CNN (0,10; 0,80; e 2,10 mmol L−1). I valori di ripristino sono presentati nella Figura S5. Nella Figura S5, è possibile osservare che il protocollo sviluppato ha fornito prestazioni analitiche soddisfacenti per il rilevamento della CNN poiché sono stati raggiunti risultati di recupero adeguati (nell’intervallo da 98,0 a 103,0% e da 95,0 a 105,0% nel siero sintetico e umano delle urine, rispettivamente).
3.3. Biosensore per COVID-19
3.3.1. Produzione di biosensori
Nella produzione del biosensore, un tempo di 400 s con 5.0 mmol L−1 è stata utilizzata una soluzione d’oro, per la quale il segnale analitico del ferrocenetanolo (sonda per l’analisi del biosensore) non presentava un segnale analitico definito (corrente di picco di ossidazione e riduzione). Pertanto, la deposizione dell’oro è stata nuovamente ottimizzata in funzione del miglior segnale analitico utilizzando il ferrocenetanolo come sonda redox. Poiché la concentrazione d’oro utilizzata in precedenza si è dimostrata una condizione ideale per questo scopo, solo il tempo di deposizione è stato ottimizzato da 100 a 300 s. L’analisi è stata eseguita mediante voltammetria ciclica in presenza di 1,0 mmol L−1 ferrocenmetanolo in 0,1 mol L−1 KCl con una velocità di scansione di 100 mV s−1. I voltammogrammi ottenuti possono essere visti nella Figura S6. Il tempo di 250 s per la deposizione di Au nello sviluppo del biosensore è stato scelto come ideale poiché presenta il più alto segnale analitico.
3.3.2. Profilo voltammetrico del biosensore proposto
Le misurazioni cicliche voltammetriche e EIS sono state eseguite prima e dopo l’immobilizzazione di una sequenza di cattura (3,0 μmol L−1 per 1 h) sulla superficie dell’Au/G-PLA utilizzando un L da 1,0 mmol−1 soluzione di ferrocenmetanolo in 0,1 mol L−1 KCl come elettrolita di supporto. I voltammogrammi ottenuti e le trame di Nyquist possono essere visti nella Figura 5.
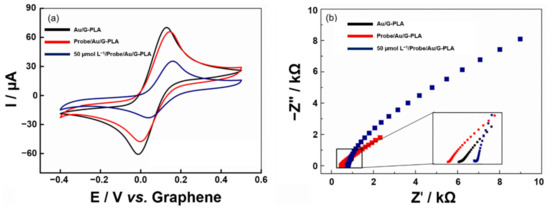
Figura 5.(a) Voltammogrammi ciclici e (b) grafici di Nyquist per ogni fase di modifica del sensore e dopo ibridazione, (linea nera) Au/G-PLA, (linea rossa) Sonda/Au/G-PLA e (linea blu) 50,0 μmol L−1 sequenza di destinazione. Tutte le analisi sono state condotte utilizzando 1,0 mmol L−1 ferrocenmetanolo in 0,1 mol L−1 KCl; I CV sono stati eseguiti con una velocità di scansione di 50 mV s−1.La Figura 5a presenta i picchi di ossidazione e riduzione della sonda redox ferrocenmetanolo su Au/G-PLA, biosensore basato su Au/G-PLA (Probe/Au/G-PLA) e biosensore in presenza di 50,0 μmol L−1 del bersaglio (Target/Sonda/Au/G-PLA). Le correnti di picco ottenute sono state di circa 65,0 e -55,0 μA per l’ossidazione e la riduzione del mediatore, rispettivamente, su Au/G-PLA. Il biosensore sviluppato ha presentato rispettivamente 62,0 e -47,0 μA, mostrando una leggera diminuzione della risposta di corrente dopo l’immobilizzazione della sequenza di cattura sulla superficie dell’elettrodo. Dopo ibridazione con 50,0 μmol L−1 della sequenza bersaglio (cDNA) del virus SARS-CoV-2, gli attuali valori di picco sono diminuiti considerevolmente (decadimento di circa il 55% nel segnale analitico) a causa di un blocco parziale dell’elettrodo da parte del materiale biologico depositato. Un comportamento simile può essere osservato anche dalle misurazioni EIS (Figura 5b) e gli attacchi sono stati eseguiti utilizzando un circuito di Randles. Un aumento di RCt valori dopo che il materiale biologico è stato depositato sulla superficie del sensore. Il sensore Au/G-PLA presentava una RCt di 200 Ω, e dal biosensore di 1,04 kΩ e dopo ibridazione con 50,0 μmol L−1 sequenza target, è stato registrato un aumento a 8,76 kΩ. Inoltre, i grafici di Bode possono essere trovati nella Figura S7. Si può notare che si è verificato lo stesso comportamento osservato negli studi CV e nelle trame di Nyquist. Gli spettri di impedenza elettrochimica aumentano mentre i cambiamenti si verificano sulla superficie del sensore stampato in 3D, principalmente nella gamma di frequenza tra 0,1 e 10 Hz, che è la regione governata dai cambiamenti nel doppio strato elettrico. Tale comportamento era atteso poiché si verifica il legame tra il cDNA di cattura e la superficie del sensore e l’ibridazione tra il cDNA di cattura e il bersaglio di interesse [65]. Pertanto, si può dedurre che il biosensore è stato assemblato con successo e risponde alla sequenza target, attestando il verificarsi del processo di ibridazione.
3.3.3. Ottimizzazione dei parametri del biosensore
I passaggi per la fabbricazione del biosensore (immobilizzazione della sequenza di cattura e ibridazione) sono stati ottimizzati per ottenere un ampio intervallo lineare e un basso LOD. Così, inizialmente, la concentrazione e il tempo di immobilizzazione della sequenza di cattura sono stati ottimizzati utilizzando la tecnica CV considerando la differenza tra l’Ibabbo ottenuto da Au/G-PLA a Probe/Au/G-PLA in presenza di 1,0 mmol L−1 ferrocenmetanolo in KCl 0,1 mol L−1. Dalle risposte presentate nella matrice CCD (Tabella S4), è stato possibile costruire la superficie di risposta e la curva di livello (Figura S8) per ottenere le migliori condizioni per la modifica del biosensore. Nella Figura S8 è stata identificata la regione di massima corrente, che è stata contrassegnata dalla “stella”, mostrando una regione stabilizzata con un intervallo di lavoro ottimale per le variabili coinvolte. I valori ottimali scelti per il biosensore sono stati 3,0 μmol L−1 e 1 ora per la concentrazione della sequenza di cattura e il tempo di deposizione, rispettivamente.In condizioni ottimali, è stato quindi studiato il tempo di ibridazione. A tale scopo, gli elettrodi sono stati incubati in una concentrazione di sequenza target di 1,0 μmol L−1 è stato incubato in periodi che vanno da 30 a 180 minuti, con l’analisi effettuata immediatamente dopo il tempo stabilito. I risultati ottenuti possono essere visti nella Figura S9, in cui si può osservare un decadimento significativo della risposta a 30 e 60 minuti. Tuttavia, da 60 minuti in poi, una diminuzione inferiore del ΔIbabbo è osservato, dimostrando che i tempi di ibridazione superiori a 60 minuti non sono efficaci. Pertanto, 30 minuti sono stati scelti come tempo di ibridazione ottimale per ulteriori studi.
3.3.4. Prestazioni analitiche del biosensore voltammetrico Signal-Off
L’applicazione del biosensore per rilevare la sequenza bersaglio del virus SARS-CoV-2 è stata studiata sulla base di una strategia di segnalazione da parte di tecniche EIS e CV in un intervallo potenziale da -0,4 a 0,5 V (vs. grafene). Il biosensore è stato testato in presenza di diverse concentrazioni della sequenza target in un intervallo da 1,0 a 50,0 e da 1,0 a 75,0 μmol L−1, rispettivamente per CV e EIS. Le correnti di picco anodiche sono state misurate in presenza di 1,0 mmol L−1 ferrocenmetanolo e 0,1 mol L−1 Kcl. Per ogni aggiunta del bersaglio della sequenza target in diverse concentrazioni sulla superficie del biosensore, la risposta della corrente anodica per il ferrocenetanolo è diminuita, il che ha indicato che il biosensore ha risposto a diverse concentrazioni del virus cDNA. Con questo, una curva analitica è stata ottenuta con le diverse tecniche (CV e EIS). I risultati ottenuti possono essere visti nella Figura 6.
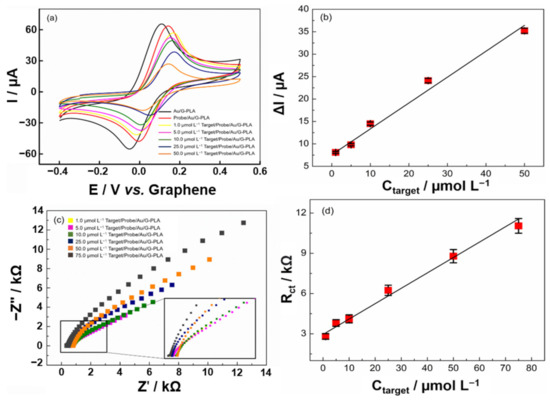
Figura 6. Curva di calibrazione del biosensore che impiega (a) tecniche CV e (c) EIS, nell’intervallo da 1,0 a 50,0 μmol L−1 e da 1,0 a 75,0 μmol L−1 target, rispettivamente. b) Sono state ottenute curve di calibrazione CV tra la differenza del segnale analitico del biosensore in assenza e presenza della sequenza target che impiega 1,0 mmol L−1 ferrocenmetanolo in 0,1 mol L−1 KCl. (d) La curva di taratura EIS è stata costruita a partire dalla R ottenutaCt valori.La curva analitica ottenuta con la tecnica CV presentava un comportamento lineare, con una R2 valore di 0,982 nell’intervallo da 1,0 a 50,0 μmol L−1. Concentrazioni più elevate del bersaglio presentavano una saturazione superficiale e non sono state osservate variazioni nella risposta corrente. L’equazione ottenuta da CV era I (μA) = 0,583 × Cbersaglio (μmol L−1) − 8,536, con una sensibilità di 0,583 μA μmol−1 L. I limiti di rilevazione e quantificazione sono stati calcolati come 0,30 e 0,95 μmol L−1rispettivamente. Le risposte ottenute da EIS hanno fornito un comportamento lineare, con una R2 valore di 0,987 nell’intervallo da 1,0 a 75,0 μmol L−1 (I grafici di Nyquist si adattano al circuito di Randles) e, a concentrazioni più elevate, è stato osservato un comportamento simile a quello ottenuto da CV. L’equazione ottenuta da EIS era RCt (kΩ) = 0,115 × Cbersaglio (μmol L−1) + 2,977, con una sensibilità di 0,115 kΩ μmol−1 L. I valori limite di rilevazione e quantificazione sono risultati essere 0,31 e 0,93 μmol L−1rispettivamente. La riproducibilità e la ripetibilità del biosensore sono state calcolate utilizzando la tecnica CV (Figura S10) e RSD dell’1,16% (n = 3; 5,0 μmol L−1), e 1,14% (n = 20; 5,0 μmol L−1) sono stati ottenuti, rispettivamente.È importante ricordare che per l’applicazione del biosensore nel rilevamento del SARS-CoV-2 in uno scenario reale, è necessario prendere in considerazione alcuni passaggi operativi. Tali passaggi sono (1) raccolta di campioni; (2) estrazione dell’RNA; (3) conversione e amplificazione del materiale genetico mediante RT-PCR; e (4) analisi del campione con il biosensore sviluppato. Tuttavia, i passaggi da 1 a 3 sono gli stessi per l’attuale protocollo di rilevamento SARS-CoV-2 basato su PCR, con il biosensore che non viene comunemente sostituito dall’elettroforesi su gel. Pertanto, il biosensore è un’alternativa all’elettroforesi su gel, mantenendo gli stessi passaggi iniziali senza aggiungere passaggi operativi e mantenendo lo stesso protocollo iniziale che è già ampiamente utilizzato [66,67]. È interessante notare che questi passaggi non sono esclusivi di questo lavoro e i biosensori elettrochimici basati sul rilevamento del cDNA sono suscettibili alla stessa procedura [11,68,69,70].Inoltre, considerando un campione con una concentrazione di materiale genetico pari al LOD del kit PCR più efficiente sul mercato (100 copie/ml), sarebbero necessari circa 40 cicli di PCR per amplificare il cDNA per raggiungere il LOD del biosensore qui sviluppato [66,67]. Ciò dimostra che, anche se l’amplificazione è necessaria, il metodo è praticabile, poiché 40 cicli richiedono circa 45 minuti per essere completati.In letteratura, alcuni lavori riportano l’uso di DNA/RNA per modificazioni di sensori elettrochimici applicati per la rilevazione del SARS-CoV-2 [28,71,72]. La Tabella S8 presenta un confronto tra i dati ottenuti dal genosensore proposto con altri presentati in letteratura per la rilevazione di SARS-CoV-2. I sensori più segnalati sono elettrodi serigrafati [71,73]. Tuttavia, l’elettrodo d’oro [72], il circuito stampato flessibilmente (a base di grafene) [74], gli elettrodi interdigitati platino / titanio su un substrato di vetro [16], oltre a un sensore stampato in 3D ottenuto utilizzando una penna di stampa 3D [28] possono anche essere trovati in letteratura. Tra questi lavori, si possono osservare le più svariate fasi di produzione dei biosensori, raggiungendo tempi superiori a 3 h. Inoltre, valori LOD che vanno da 33,0 fmol L−1 a 0,1 μmol L−1 sono osservati. Tuttavia, vale la pena ricordare che alcuni studi utilizzano materiali ad alto costo, come platino, titanio e oro, soprattutto se confrontati con i materiali utilizzati nel presente lavoro (principalmente grafene, PLA e sale d’oro). Inoltre, l’uso della stampa 3D è scarsamente esplorato, con un solo lavoro che utilizza una penna di stampa 3D in combinazione con una sonda ssDNA che ha preso di mira la sequenza del gene N di SARS-CoV-2. Pertanto, il presente lavoro presenta un nuovo approccio alla stampa 3D che consente la produzione rapida e automatizzata di sensori miniaturizzati, riducendo i costi operativi e promuovendo la facilità d’uso, e per lo sviluppo di un genosensore per cDNA di SARS-CoV-2.
3.3.5. Studio di interferenza
Infine, il biosensore è stato valutato in presenza di un bersaglio negativo (sequenza di DNA dell’influenza A) per osservare la selettività del metodo. La figura 7 presenta i voltammogrammi ciclici ottenuti.
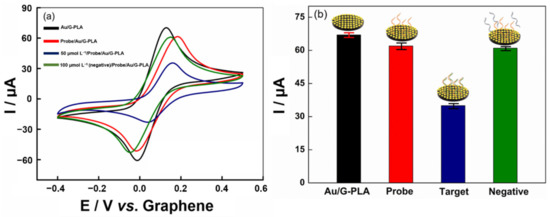
Figura 7.a) Confronto tra sequenze target positive (50 μmol L−1) e negativo (100 μmol L−1). Voltammogrammi ciclici ottenuti in presenza di 1,0 mmol L−1 ferrocenmetanolo in 0,1 mol L−1 Kcl. La velocità di scansione era di 100 mV s−1, b) Grafico a colonne a barre.Come previsto, la sequenza di DNA di controllo negativo non mostra alcun cambiamento significativo nel segnale analitico rispetto al segnale del biosensore. D’altra parte, l’aggiunta di 50,0 μmol L−1 della sequenza target ha causato un forte calo della corrente di picco (circa il 50%), poiché le sequenze target si sono ibridate con la sequenza di cattura (sonda). Questo è un tasso più alto del controllo delle sequenze negative, poiché il controllo delle sequenze negative non ha la specificità necessaria per l’ibridazione. Questo comportamento è attribuito alla sua complementarità con la sequenza di acquisizione. Pertanto, una sequenza di DNA dal virus può essere rilevata con una buona specificità. Inoltre, il virus dell’influenza fornisce sintomi simili in un individuo infetto a SARS-CoV-2; pertanto, il DNA di questo virus è stato scelto per essere utilizzato come sequenza negativa.Infine, per dimostrare l’applicabilità del biosensore e del sensore, l’analisi di Target è stata eseguita su saliva sintetica e siero umano mediante test di recupero dalla fortificazione di campioni con tre diverse concentrazioni di Target (1,0, 25,0 e 50,0 μmol L−1). Le risposte analitiche sono state ottenute dopo 30 minuti di incubazione dei campioni sulla superficie del biosensore stampato in 3D e le risposte dei valori di recupero (espressi in %) possono essere viste nella Figura S11. I valori di recupero ottenuti variavano dal 96,0 al 102,0% per la saliva sintetica e dal 97,0 al 101,0% per il siero umano. Pertanto, il biosensore proposto si rivela adatto per l’analisi del campione, avendo un buon potenziale per determinare le caratteristiche del virus SARS-CoV-2, in modo semplice, rapido e utilizzando materiali a basso costo.
4. Conclusioni
Qui, abbiamo presentato lo sviluppo di sensori (bio)stampati in 3D modificati da Au con un grande potenziale per il rilevamento di SARS-CoV-2, il virus responsabile di COVID-19 e un possibile biomarcatore per la malattia, la CNN. La strategia di rilevazione impiegata per la costruzione dei dispositivi analitici si è basata sulla risposta di spegnimento del segnale per il metallo Au presente sulla superficie di un elettrodo stampato in 3D, dopo la complessazione con CNN per il sensore e dopo l’interazione con il filamento di DNA complementare del virus (SARS-CoV-2) immobilizzato sulla superficie del sensore Au con una sequenza target (filamento di DNA complementare del virus SARS-CoV-2) per il biosensore. Il rilevamento CNN è stato eseguito con successo, consentendo il rilevamento di basse concentrazioni di questo analita (LOD = 0,016 mmol L-1) con alta sensibilità (9,318 μA mmol−1 L) in campioni sintetici di urina e siero umano, fornendo adeguati valori di recupero, che variavano dal 94,0 al 110,0%. L’immobilizzazione del materiale biologico sulla piattaforma sviluppata ha fornito la fabbricazione di un biosensore rapido per il rilevamento di SARS-CoV-2, richiedendo solo un tempo di ibridazione di 30 minuti, con un basso LOD (0,30 μmol L−1) e sensibilità soddisfacente (0,583 μA μmol−1 L) in campioni di saliva sintetica e siero umano, fornendo adeguati valori di recupero, che variavano dal 95,0 al 105,0%. È stata ottenuta un’elevata precisione nella fabbricazione dei biosensori (RSD = 1,16%) e sono state osservate misurazioni riproducibili (RSD < 1,16%) senza interferenze del materiale genetico del virus dell’influenza A. Pertanto, il rilevamento di SARS-CoV-2 è stato eseguito utilizzando una piattaforma semplice e versatile, utilizzando la tecnologia di stampa 3D. Anche tenendo conto della necessità di amplificazione del cDNA mediante PCR, questo passaggio può ancora essere considerato veloce, come discusso in precedenza, portando fattibilità e potenziale di applicazione in uno scenario reale.Inoltre, lo sviluppo di un test elettrochimico rapido per COVID-19, combinato con la determinazione di un biomarcatore, fornirebbe risultati più informativi, come l’aggressività della malattia, consentendo una migliore gestione e trattamento in una popolazione infetta. Infine, la piattaforma stampata in 3D ha il vantaggio di miniaturizzazione, facilità di produzione, costo relativamente basso e facile gestione.
5. Brevetti
Il presente lavoro ha generato un brevetto intitolato “Biosensore elettrochimico stampato in 3D per il rilevamento del virus COVID-19” con numero d’ordine: BR 10 2021 018602 0 alla data del 17 settembre 2021.
Materiali supplementari
Le seguenti informazioni di supporto possono essere scaricate all’indirizzo: https://www.mdpi.com/article/10.3390/bios12080622/s1: Tabella S1. Matrice di pianificazione CCD, livelli reali e normalizzati e risposta ottenuta per Au/G-PLA. Tabella S2. Matrice di progettazione fattoriale completa (23) e i rispettivi risultati ottenuti per la CNN. Tabella S3. Matrice di pianificazione CCD, livelli reali e normalizzati e risposta per CNN. Tabella S4. Matrice di pianificazione CCD, livelli reali e normalizzati e risposta per SARS-CoV-2. Tabella S5. Risultato degli effetti calcolati per la pianificazione 23. Tabella S6. Elettrodi trovati in letteratura per la determinazione della creatinina. Tabella S7. Effetto di possibili especie di interferenza sulla determinazione della CNN. Tabella S8. Confronto tra le caratteristiche del genosensore proposte e le opere della letteratura. Schema S1. Illustrazione schematica dell’assemblaggio di elettrodi stampati in 3D. Fissaggio sul supporto in fogli di plastica dura e delimitazione dell’area con smalto incolore. Figura S1. Spettro EDX per Au/G-PLA. La barra della scala corrisponde a 50 μm. Figura S2. Analisi di impedenza degli elettrodi G-PLA e Au/G-PLA. a) correlazione di entità e frequenza dell’impedenza del G-PLA (linea nera) e dell’Au/G-PLA (linea rossa), b) sfasamento e correlazione di frequenza del G-PLA (linea nera) e dell’Au/G-PLA (linea rossa), c) diagrammi di Nyquist del G-PLA (linea nera) e Au/G-PLA (linea rossa). Parametri: EG-PLA = 0,6 mV; EAu/G-PLA = 9,0 mV. L’inserto mostra i rispettivi circuiti equivalenti. Figura S3. Voltammogrammi ciclici ottenuti per 1,0 mmol L−1 ferrocenmetanolo in 0,1 mol L−1 KCl che varia la velocità di scansione in 20, 40, 60, 80, 100, 120, 140, 160, 180 e 200 mV s−1 utilizzando (a) G-PLA e (c) Au/G-PLA e il rispettivo grafico della risposta corrente in funzione di v1/2 per (b) G-PLA e (d) Au/G-PLA. Figura S4. (a) Risposta superficiale e (b) curva di livello ottenuta per l’ottimizzazione delle variabili: potenziale di passo e frequenza in funzione della corrente in presenza di 3,0 mmol L−1 Cnn. Figura S5. Test di recupero eseguito dalla fortificazione di campioni di (a) urina sintetica e (b) siero umano con tre concentrazioni note di CNN (0,1, 0,8 e 2,1 mmol L−1). Figura S6. Voltammogrammi ciclici ottenuti dopo diversi tempi di deposizione dell’oro sul sensore G-PLA. (a) (linea nera) 100 s, (linea rossa) 150 s, (linea blu) 200, (linea rosa) 250 e (linea verde) 300. Tutte le analisi sono state effettuate utilizzando 1,0 mmol L−1 ferrocenmetanolo in 0,1 mol L−1 KCl con una velocità di scansione di 50 mV s−1. Figura S7. Analisi dell’impedenza del (linea nera) Au/G-PLA, (linea rossa) Sonda/Au/G-PLA e (linea blu) 50,0 μmol L-1 Target/Sonda/Au/G-PLA. a) entità dell’impedenza e correlazione di frequenza. b) sfasamento e correlazione di frequenza. Figura S8. (a) Risposta superficiale e (b) curva di livello ottenuta per l’ottimizzazione delle variabili: concentrazione (μmol L−1) e tempo (h) in funzione della differenza di corrente tra il segnale analitico del sensore e il biosensore in presenza di 1,0 mmol L−1 fer-rocenemetanolo in 0,1 mol L−1 KCl. Figura S9. Risposte ottenute per tempi diversi (min) di ibridazione della sequenza target con il biosensore. I valori di risposta sono stati ottenuti in funzione della differenza nel segnale ottenuto per CV tra il biosensore in assenza e la presenza di 1,0 μmol L−1 di sequenza target con 1 mmol L−1 ferrocenmetanolo in 0,1 mol L−1 I CV KCl. sono stati eseguiti con una velocità di scansione di 50 mV s−1. Figura S10. Voltammogrammi ciclici per (a) riproducibilità del biosensore (n = 3) e (b) ripetibilità (n = 20) in presenza di 1,0 mmol L−1 ferrocenmetanolo in 0,1 mol L−1 KCl. Velocità di scansione di 100 mV s−1. Figura S11. Test di recupero eseguito dalla fortificazione di campioni (a) urina sintetica e (b) siero umano con tre concentrazioni di Target (1,0, 25,0 e 50,0 μmol L−1). Video S1: Preparazione del biosensore. I riferimenti [75,76,77] sono citati nei materiali supplementari.
Contributi dell’autore
L.R.G.S.: Conceptualization, Investigation, Validation, Methodology, Visualization, Data curation, Writing—original draft preparation, Writing—review and editing. J.S.S.: Conceptualization, Investigation, Validation, Methodology, Visualization, Data curation, Writing—original draft preparation, Writing—original draft. L.O.O.: Conceptualization, Investigation, Visualization, Data curation, Writing—review and editing. L.C.B.: Investigation, Visualization, Methodology, Data curation, Writing—review and editing. E.C.: Visualization, Funding acquisition, Writing—review and editing. L.H.M.-J.: Conceptualization, Visualization, Funding acquisition, Writing—review and editing. M.F.B.: Conceptualization, Visualization, Funding acquisition, Writing—review and editing. R.A.A.M.: Conceptualization, Visualization, Writing—review and editing. B.C.J.: Conceptualization, Visualization, Supervision, Project administration, Resources, Funding acquisition, Writing—review and editing. All authors have read and agreed to the published version of the manuscript.
Funding
This research was funded by FAPESP, grant number 2017/21097-3, 2022/06145-0 and 2018/19750-3, CAPES, grant number 001, 88887.510506/2020-00 and 88887.510880/2020-00, CNPq, grant number 303338/2019-9, 427731/2018-6, and 307271/2017-0 and INCTBio (CNPq), grant number 465389/2014–7.
Institutional Review Board Statement
Not applicable.
Informed Consent Statement
Not applicable.
Data Availability Statement
Not applicable.
Riconoscimenti
Gli autori sono grati alle agenzie brasiliane FAPESP (2017/21097-3, 2022/06145-0 e 2018/19750-3), CAPES (001), CAPES (88887.510506/2020-00 e 88887.510880/2020-00), CNPq (303338/2019-9, 427731/2018-6 e 307271/2017-0) e INCTBio (sovvenzione CNPq n. 465389/2014-7) per il sostegno finanziario.
Conflitti di interesse
Gli autori non dichiarano alcun conflitto di interessi. I finanziatori non hanno avuto alcun ruolo nella progettazione dello studio; nella raccolta, analisi o interpretazione dei dati; nella stesura del manoscritto o nella decisione di pubblicare i risultati.
Referenze
- Stefano, J.S.; Kalinke, C.; da Rocha, R.G.; Rocha, D.P.; da Silva, V.A.O.P.; Bonacin, J.A.; Angnes, L.; Richter, E.M.; Janegitz, B.C.; Muñoz, R.A.A. Sensori elettrochimici (bio)abilitati dalla stampa 3D basata sulla modellazione a deposizione fusa: una guida alla selezione di progetti, parametri di stampa e protocolli di post-trattamento. Chem. 2022, 94, 6417–6429. [Google Scholar] [CrossRef]
- Liyarita, B.R.; Ambrosi, A.; Pumera, M.3D elettrodi stampati per il rilevamento di molecole biologicamente attive. Wiley Online Libr. 2018, 30, 1319–1326. [Google Scholar] [CrossRef]
- Cardoso, R.M.; Kalinke, C.; Rocha, R.G.; dos Santos, P.L.; Rocha, D.P.; Oliveira, P.R.; Janegitz, B.C.; Bonacin, J.A.; Richter, E.M.; Munoz, R.A.A. Sensori elettrochimici prodotti in additivo (stampati in 3D): una revisione critica. Chim. Acta 2020, 1118, 73–91. [Google Scholar] [CrossRef] [PubMed]
- Whittingham, M.J.; Crapnell, R.D.; Rothwell, E.J.; Hurst, N.J.; Banks, C.E. Additive manufacturing for electrochemical labs: An overview and tutorial note on the production of cells, electrodes and accessories. Talanta Open 2021, 4, 100051. [Google Scholar] [CrossRef]
- Jayaprakash, G.K. Pre-post redox electron transfer regioselectivity at the alanine modified nano graphene electrode interface. Chem. Phys. Lett. 2022, 789, 139295. [Google Scholar] [CrossRef]
- Coroş, M.; Pruneanu, S.; Stefan-van Staden, R.-I. Recenti progressi nei sensori elettrochimici e nei biosensori a base di grafene. J. Elettrochimico Soc. 2020, 167, 037528. [Google Scholar] [CrossRef]
- Wisitsoraat, A.; Tuantranont, A. Chimica e biosensori a base di grafene; Springer: Berlino/Heidelberg, Germania, 2013; pp. 103-141. [Google Scholar]
- Foster, C.W.; Giù, M.P.; Zhang, Sì; Ji, X.; Rowley-Neale, S.J.; Smith, G.C.; Kelly, P.J.; Banks, C.E.3D Dispositivi di accumulo di energia basati sul grafene stampato. 2017, 7, 42233. [Google Scholar] [CrossRef]
- Abdalla, A.; Patel, B.A.3D-printed electrochemical sensors: A new horizon for measurement of biomolecules. Curr. Opin. Elettrochimica. 2020, 20, 78–81. [Google Scholar] [CrossRef]
- Muñoz, J.; Immunosensori COVID-19 pumera, M.3D-stampati con lettura elettronica. Chem. Ing. J. 2021, 425, 131433. [Google Scholar] [CrossRef]
- Khan, M.Z.H.; Hasan, M.R.; Hossain, S.I.; Ahommed, M.S.; Daizy, M. Rilevamento ultrasensibile di virus patogeni con biosensore elettrochimico: stato dell’arte. Biosens. Bioelettrone. 2020, 166, 112431. [Google Scholar] [CrossRef]
- Malik, Stati Uniti; Kumar, N.; Sircar, S.; Kaushik, R.; Bhat, S.; Dhama, K.; Gupta, P.; Goyal, K.; Singh, M.P.; Ghoshal, Stati Uniti; et al. Pandemia di coronavirus (COVID-19): sfide e una prospettiva globale. Agenti patogeni 2020, 9, 519. [Google Scholar] [CrossRef]
- Vandenberg, O.; Martiny, D.; Rochas, O.; van Belkum, A.; Kozlakidis, Z. Considerazioni per i test diagnostici COVID-19. Nat. Rev. Microbiolo. 2021, 19, 171–183. [Google Scholar] [CrossRef]
- Giri, B.; Pandey, S.; Shrestha, R.; Pokharel, K.; Ligler, F.S.; Neupane, B.B. Revisione delle prestazioni analitiche dei metodi di rilevamento COVID-19. Anal. Bioanal. Chem. 2021, 413, 35–48. [Google Scholar] [CrossRef]
- Corman, V.M.; Landt, O.; Kaiser, M.; Molenkamp, R.; Meijer, A.; Chu, D.K.W.; Bleicker, T.; Brünink, S.; Schneider, J.; Schmidt, M.L.; et al. Rilevamento del nuovo coronavirus 2019 (2019-nCoV) mediante RT-PCR in tempo reale. Eurosurveglianza 2020, 25, 2000045. [Google Scholar] [CrossRef]
- Hwang, C.; Parco, N.; Kim, E.S.; Kim, M.; Kim, S.D.; Parco, S.; Kim, New York; Kim, J.H. Biosensore di DNA ultraveloce e riciclabile per il rilevamento point-of-care di SARS-CoV-2 (COVID-19). Biosens. Bioelettrone. 2021, 185, 113177. [Google Scholar] [CrossRef]
- Brazaca, L.C.; Bramorski, C.B.; Cancino-Bernardi, J.; Janegitz, B.C.; Zucolotto, V. Un genosensore per la determinazione del tratto dell’anemia falciforme. Wiley Online Libr. 2017, 29, 773–777. [Google Scholar] [CrossRef]
- Beduk, T.; Beduk, D.; de Oliveira Filho, J.I.; Zihnioglu, F.; Cicek, C.; Sertoz, R.; Arda, B.; Goksel, T.; Turhan, K.; Salama, K.N.; et al. Diagnosi rapida di COVID-19 point-of-care con un biosensore di grafene laser-scribato assistito da nanoarchitetture dorate. Chem. 2021, 93, 8585–8594. [Google Scholar] [CrossRef]
- Peng, Y.; Pan, Y.; Sole, Z.; Li, J.; Yi, Sì; Yang, J.; Li, G. Un biosensore elettrochimico per l’analisi sensibile dell’RNA SARS-CoV-2. Biosens. Bioelettrone. 2021, 186, 113309. [Google Scholar] [CrossRef]
- Wang, M.; Lin, Y.; Lu, J.; Sole, Z.; Deng, Y.; Wang, L.; Yi, Sì; Li, J.; Yang, J.; Li, G. Rilevamento visivo ad occhio nudo dell’RNA SARS-CoV-2 basato su capsule organiche covalenti. Chem. Ing. J. 2022, 429, 132332. [Google Scholar] [CrossRef]
- Ghanbari, K.; Roushani, M.; Azadbakht, A. Aptasensor ultrasensibile basato su un nanocomposito GQD per il rilevamento dell’antigene del nucleo del virus dell’epatite C. Anal. Biochimica. 2017, 534, 64–69. [Google Scholar] [CrossRef]
- Li, X.; Scida, K.; Crooks, R.M. Rilevamento del DNA del virus dell’epatite B con un sensore elettrochimico di carta. Chem. 2015, 87, 9009–9015. [Google Scholar] [CrossRef]
- Brazaca, L.C.; Imamura, A.H.; Gomes, N.O.; Almeida, M.B.; Scheidt, D.T.; Raymundo-Pereira, P.A.; Oliveira, N.O.; Janegitz, B.C.; Machado, S.A.S.; Carrilho, E. Immunosensori elettrochimici che utilizzano nanostrutture d’oro elettrodepositate per rilevare le proteine S da SARS-CoV e SARS-CoV-2. Anal. Bioanal. Chem. 2022, 414, 5507–5517. [Google Scholar] [CrossRef]
- Stefano, J.S.; Guterres e Silva, L.R.; Rocha, R.G.; Brazaca, L.C.; Richter, E.M.; Abarza Muñoz, R.A.; Janegitz, B.C. Nuovo filamento conduttivo pronto all’uso per la stampa 3D di sensori elettrochimici (bio): verso il rilevamento di SARS-CoV-2. Chim. Acta 2021, 1191, 339372. [Google Scholar] [CrossRef]
- Brazaca, L.C.; dos Santos, P.L.; de Oliveira, P.R.; Rocha, D.P.; Stefano, J.S.; Kalinke, C.; Abarza Muñoz, R.A.; Bonacin, J.A.; Janegitz, B.C.; Carrilho, E. Strategie di biorilevamento per il rilevamento elettrochimico di virus e malattie virali : una recensione. Chim. Acta 2021, 1159, 338384. [Google Scholar] [CrossRef]
- Tripatia, S.; Singh, S.G. Rilevamento elettrochimico senza etichette dell’ibridazione del DNA: un metodo per la diagnosi di COVID-19. Trans. Indian Natl. Acad. 2020, 5, 205–209. [Google Scholar] [CrossRef]
- Shi, L.; Wang, L.; Ma, X.; Zanna, X.; Xiang, L.; Yi, Sì; Li, J.; Luo, Z.; Li, G. Nanocanali funzionalizzati con Aptamer per il rilevamento in un’unica fase di SARS-CoV-2 in campioni di pazienti COVID-19. Chem. 2021, 93, 16646–16654. [Google Scholar] [CrossRef]
- Crevillen, A.G.; Mayorga-Martinez, C.C.; Vaghasiya, J.V.; Pumera, M.3D-printed SARS-CoV-2 RNA sistema microfluidico genosensante. Mater. Technol. 2022, 7, 2101121. [Google Scholar] [CrossRef]
- Werion, A.; Belkhir, L.; Perrot, M.; Schmit, G.; Aydin, S.; Chen, Z.; Penaloza, A.; De Greef, J.; Yildiz, H.; Pothen, L.; ET AL. SARS-CoV-2 provoca una disfunzione specifica del tubulo prossimale renale. Rene Int. 2020, 98, 1296–1307. [Google Scholar] [CrossRef]
- Xiang, J.; Wen, J.; Yuan, X.; Xiong, S.; Zhou, X.; Liu, C.; Min, X. Potenziali marcatori biochimici per identificare i casi gravi tra i pazienti COVID-19. medRxiv 2020. [Google Scholar] [CrossRef]
- Li, T.J.; Chen, P.Y.; Nien, P.C.; Lin, C.Y.; Vittal, R.; Ling, T.R.; Ho, K.C. Preparazione di un nuovo polimero a impronta molecolare mediante il processo sol-gel per il rilevamento della creatinina. Chim. Acta 2012, 711, 83–90. [Google Scholar] [CrossRef]
- Jacobi, D.; Lavigne, C.; Halimi, J.M.; Fierrard, H.; Andres, C.; Couet, C.; Maillot, F. Variabilità nell’escrezione di creatinina in uomini e donne diabetici adulti, in sovrappeso: conseguenze sulla classificazione della malattia renale basata sulla creatinina. Diabete Res. Prassi. 2008, 80, 102–107. [Google Scholar] [CrossRef] [PubMed]
- Lad, U.; Khokhar, S.; Kale, G.M. Biosensori elettrochimici della creatinina. Chem. 2008, 80, 7910–7917. [Google Scholar] [CrossRef] [PubMed]
- Vescovo, M.L.; Fody, E.P.; Schoeff, L.E. Clinical Chemistry: Principles, Techniques, and Correlations, 7th ed.; Wolters Kluwer Health/Hippincott Williams & Wilkins: Philadelphia, PA, USA, 2013; ISBN 9781469837130. [Google Scholar]
- Brutis, C.A.; Bruns, D.E. Tietz Fundamentals of Clinical Chemistry and Molecular Diagnostics, 7a ed.; Elsevier-Health Sciences Division: St. Louis, MO, USA, 2014; ISBN 9780323292061. [Google Scholar]
- Vella, F. Libro di testo di chimica clinica. Biochimica. 1986, 14, 146. [Google Scholar] [CrossRef]
- Chan, K.H.; Farouji, I.; Abu Hanoud, A.; Slim, J. Debolezza ed elevata creatinina chinasi come presentazione iniziale della malattia da coronavirus 2019 (COVID-19). Am. J. Emerg. 2020, 38, 1548.e1–1548.e3. [Google Scholar] [CrossRef]
- Stradiotto, N.R.; Yamanaka, H.; Zanoni, M.V.B. Sensori elettrochimici: un potente strumento in chimica analitica. J. Braz. Chem. Soc. 2003, 14, 159–173. [Google Scholar] [CrossRef]
- Fare, J.S.; Chang, Y.H.; Tsai, M.L. Biosensore amperometrico di creatinina altamente sensibile basato su film di rilevamento composito di polianilina creatinina deiminasi/Nafion-nanostrutturato preparato con voltammetria ciclica. Mater. Chem. Phys. 2018, 219, 1–12. [Google Scholar] [CrossRef®]
- Fava, E.L.; Prado, T.M.D.; Garcia-Filho, A.; Silva, T.A.; Cincotto, F.H.; Cruz de Moraes, F.; Faria, R.C.; Fatibello-Filho, O. Determinazione elettrochimica non enzimatica della creatinina utilizzando una nuova microcella serigrafata. Talanta 2020, 207, 120277. [Google Scholar] [CrossRef]
- Kumar, V.; Hebbar, S.; Kalam, R.; Panwar, S.; Prasad, S.; Srikanta, S.S.; Krishnaswamy, P.R.; Bhat, N. Creatinine-iron complex and its use in electrochemical measurement of urine creatinine. IEEE Sens. J. 2018, 18, 830–836. [Google Scholar] [CrossRef]
- Naresh Kumar, T.; Ananthi, A.; Mathiyarasu, J.; Joseph, J.; Lakshminarasimha Phani, K.; Yegnaraman, V. Enzymeless creatinine estimation using poly(3,4-ethylenedioxythiophene)-β-cyclodextrin. J. Electroanal. Chem. 2011, 661, 303–308. [Google Scholar] [CrossRef]
- Hooshmand, S.; Es’haghi, Z. Microfabricated disposable nanosensor based on CdSe quantum dot/ionic liquid-mediated hollow fiber-pencil graphite electrode for simultaneous electrochemical quantification of uric acid and creatinine in human samples. Anal. Chim. Acta 2017, 972, 28–37. [Google Scholar] [CrossRef]
- Randviir, E.P.; Brownson, D.A.C.; Banks, C.E. A decade of graphene research: Production, applications and outlook. Mater. Today 2014, 17, 426–432. [Google Scholar] [CrossRef]
- Raveendran, J.; Resmi, P.E.; Ramachandran, T.; Nair, B.G.; Satheesh Babu, T.G. Fabrication of a disposable non-enzymatic electrochemical creatinine sensor. Sens. Actuators B Chem. 2017, 243, 589–595. [Google Scholar] [CrossRef]
- Rakesh Kumar, R.K.; Shaikh, M.O.; Chuang, C.H. A review of recent advances in non-enzymatic electrochemical creatinine biosensing. Anal. Chim. Acta 2021, 1183, 338748. [Google Scholar] [CrossRef]
- Cardoso, R.M.; Castro, S.V.F.; Silva, M.N.T.; Lima, A.P.; Santana, M.H.P.; Nossol, E.; Silva, R.A.B.; Richter, E.M.; Paixão, T.R.L.C.; Muñoz, R.A.A. 3D-printed flexible device combining sampling and detection of explosives. Sens. Actuators B Chem. 2019, 292, 308–313. [Google Scholar] [CrossRef]
- Ngo, T.D.; Kashani, A.; Imbalzano, G.; Nguyen, K.T.Q.; Hui, D. Additive manufacturing (3D printing): A review of materials, methods, applications and challenges. Compos. Part B Eng. 2018, 143, 172–196. [Google Scholar] [CrossRef]
- Da Silva, V.A.O.P.; Tartare, V.A.P.; Kalinke, C.; Roberto De Oliveira, P.; Cardoso De Souza, D.; Bonacin, J.A.; Janegitz, B.C. Construção de um suporte ajustável lab-made impresso em 3D para medição de ângulo de contato. Quim. Nov. 2020, 43, 1312–1319. [Google Scholar] [CrossRef]
- Guo, S.; Wang, L.; Wang, E. Templateless, surfactantless, simple electrochemical route to rapid synthesis of diameter-controlled 3D flowerlike gold microstructure with “clean” surface. Chem. Commun. 2007, 3163–3165. [Google Scholar] [CrossRef]
- Sun, X.; Dong, S.; Wang, E. Large-scale synthesis of micrometer-scale single-crystalline Au plates of nanometer thickness by a wet-chemical route. Angew. Chem. Int. Ed. 2004, 43, 6360–6363. [Google Scholar] [CrossRef]
- Zhang, J.; Du, J.; Han, B.; Liu, Z.; Jiang, T.; Zhang, Z. Sonochemical formation of single-crystalline gold nanobelts. Wiley Online Libr. 2006, 45, 1116–1119. [Google Scholar] [CrossRef]
- Antonini, S.; Nguyen, P.T.; Arnold, U.; Eichert, T.; Clemens, J. Solar thermal evaporation of human urine for nitrogen and phosphorus recovery in Vietnam. Sci. Total Environ. 2012, 414, 592–599. [Google Scholar] [CrossRef]
- Romonti, D.E.; Gomez Sanchez, A.V.; Milošev, I.; Demetrescu, I.; Ceré, S. Effect of anodization on the surface characteristics and electrochemical behaviour of zirconium in artificial saliva. Mater. Sci. Eng. C 2016, 62, 458–466. [Google Scholar] [CrossRef]
- Barros Neto, B.; Scarminio, I.S.; Bruns, R.E. Como Fazer Experimentos: Pesquisa e Desenvolvimento na Ciência e na Indústria; Editora da Unicamp: Campinas, SP, Brazil, 2001. [Google Scholar]
- Teófilo, R.F.; Ferreira, M.M.C. Chemometrics II: Spreadsheets for experimental design calculations, A tutorial. Quim. Nova 2006, 29, 338–350. [Google Scholar] [CrossRef]
- Yi, M.; Zhang, C. The synthesis of two-dimensional MoS2 nanosheets with enhanced tribological properties as oil additives. RSC Adv. 2018, 8, 9564–9573. [Google Scholar] [CrossRef]
- Yagati, A.K.; Go, A.; Vu, N.H.; Lee, M.H. A MoS2–Au nanoparticle-modified immunosensor for T3 biomarker detection in clinical serum samples. Electrochim. Acta 2020, 342, 136065. [Google Scholar] [CrossRef]
- Chi, H.; Liu, B.; Guan, G.; Zhang, Z.; Han, M.Y. A simple, reliable and sensitive colorimetric visualization of melamine in milk by unmodified gold nanoparticles. Analyst 2010, 135, 1070–1075. [Google Scholar] [CrossRef]
- Cui, H.; Wang, W.; Duan, C.F.; Dong, Y.P.; Guo, J.Z. Synthesis, characterization, and electrochemiluminescence of luminolreduced gold nanoparticles and their application in a hydrogen peroxide sensor. Chem.—A Eur. J. 2007, 13, 6975–6984. [Google Scholar] [CrossRef]
- He, Y.; Peng, R. Luminol functionalized gold nanoparticles as colorimetric and chemiluminescent probes for visual, label free, highly sensitive and selective detection of minocycline. Nanotechnology 2014, 25, 455502. [Google Scholar] [CrossRef]
- Gittins, D.I.; Caruso, F. Spontaneous phase transfer of nanoparticulate metals from organic to aqueous media. Angew. Chemie—Int. Ed. 2001, 40, 3001–3004. [Google Scholar] [CrossRef]
- He, Y.; Zhang, X.; Yu, H. Gold nanoparticles-based colorimetric and visual creatinine assay. Microchim. Acta 2015, 182, 2037–2043. [Google Scholar] [CrossRef]
- Ziegel, E.R.; Massart, D.L.; Vandeginste, B.G.M.; Buydens, L.M.C.; de Jong, S.; Lewi, P.J.; Verbeke, J.S. Handbook of chemometrics and qualimetrics, part B. Technometrics 2000, 42, 218. [Google Scholar] [CrossRef]
- Carr, O.; Raymundo-Pereira, P.A.; Shimizu, F.M.; Sorroche, B.P.; Melendez, M.E.; de Oliveira Pedro, R.; Miranda, P.B.; Carvalho, A.L.; Reis, R.M.; Arantes, L.M.R.B.; et al. Genosensor made with a self-assembled monolayer matrix to detect MGMT gene methylation in head and neck cancer cell lines. Talanta 2020, 210, 120609. [Google Scholar] [CrossRef] [PubMed]
- Arnaout, R.; Lee, R.A.; Lee, G.R.; Callahan, C.; Yen, C.F.; Smith, K.P.; Arora, R.; Kirby, J.E. SARS-CoV2 testing: The limit of detection matters. bioRxiv 2020. [Google Scholar] [CrossRef]
- Bustin, S.A. How to speed up the polymerase chain reaction. Biomol. Detect. Quantif. 2017, 12, 10–14. [Google Scholar] [CrossRef] [PubMed]
- Yu, H.L.L.; Maslova, A.; Hsing, I.-M. Rational design of electrochemical DNA biosensors for point-of-care applications. ChemElectroChem 2017, 4, 795–805. [Google Scholar] [CrossRef]
- Dubé, C.; Kelton, D.F.; Dubé, C.; Ribble, C.; Kelton, D.; Mcnab, B. Introduction to network analysis and its implications for animal disease modelling. Rev. Sci. Tech. Off. Int. Epiz. 2011, 30, 425–436. [Google Scholar] [CrossRef]
- Janegitz, B.C.; Cancino, J.; Zucolotto, V. Disposable biosensors for clinical diagnosis. J. Nanosci. Nanotechnol. 2014, 14, 378–389. [Google Scholar] [CrossRef]
- del Caño, R.; García-Mendiola, T.; García-Nieto, D.; Álvaro, R.; Luna, M.; Iniesta, H.A.; Coloma, R.; Diaz, C.R.; Milán-Rois, P.; Castellanos, M.; et al. Amplification-free detection of SARS-CoV-2 using gold nanotriangles functionalized with oligonucleotides. Microchim. Acta 2022, 189, 171. [Google Scholar] [CrossRef]
- Deng, Y.; Peng, Y.; Wang, L.; Wang, M.; Zhou, T.; Xiang, L.; Li, J.; Yang, J.; Li, G. Target-triggered cascade signal amplification for sensitive electrochemical detection of SARS-CoV-2 with clinical application. Anal. Chim. Acta 2022, 1208, 339846. [Google Scholar] [CrossRef]
- Ang, W.L.; Lim, R.R.X.; Ambrosi, A.; Bonanni, A. Rilevamento elettrochimico rapido della sequenza genomica COVID-19 con biosensore basato su nanocolloidi di grafene a doppia funzione. FlatChem 2022, 32, 100336. [Google Scholar] [CrossRef]
- Damiati, S.; Sopstad, S.; Pavone, M.; Akhtar, A.S.; Pinto, I.; Soares, R.R.G.; Russom, A. Il circuito stampato Flex ha implementato un aensor del DNA a base di grafene per la setezione di SARS-CoV-2. IEEE Sens. 2021, 21, 13060–13067. [Google Scholar] [CrossRef]
- Kozitsina, A.N.; Shalygina, Z.V.; Dedeneva, S.S.; Rusinov, G.L.; Tolshchina, S.G.; Verbitskiy, E.V.; Brainina, K.Z. Sistemi catalitici basati sui complessi di nichel organico (II) nella determinazione cronoamperometrica dell’urea e della creatinina. Russ. Chem. Bull. 2009, 58, 1119–1125. [Google Scholar] [CrossRef]
- Chen, J.C.; Kumar, A.S.; Chung, H.H.; Chien, S.H.; Kuo, M.C.; Zen, J.M. Un sensore elettrochimico senza enzimi per la determinazione selettiva della creatinina nelle urine umane. Attuatori B Chem. 2006, 115, 473–480. [Google Scholar] [CrossRef]
- Randviir, E.P.; Kampouris, D.K.; Banche, C.E. Un metodo di rilevamento della creatinina elettrochimica migliorato tramite una procedura basata su Jaffe. Analista 2013, 138, 6565–6572. [Google Scholar] [CrossRef]
| Nota dell’editore: MDPI rimane neutrale per quanto riguarda le rivendicazioni giurisdizionali nelle mappe pubblicate e nelle affiliazioni istituzionali. |
© 2022 dagli autori. Licenziatario MDPI, Basilea, Svizzera. Questo articolo è un articolo ad accesso aperto distribuito secondo i termini e le condizioni della licenza Creative Commons Attribution (CC BY) (https://creativecommons.org/licenses/by/4.0/).