
DIPARTIMENTO DI MEDICINA MOLECOLARE E TRASLAZIONALE
SEZIONE DI MICROBIOLOGIA
UNIVERSITA’ DI BRESCIA
Prof. Arnaldo Caruso – Presidente Società Italiana di Virologia
“Valutazione comparativa della sensibilità e specificità del sistema di diagnosi molecolare rapida per SARS-CoV-2 Shineway”

INTRODUZIONE e RATIONALE
Nel dicembre 2019 un importante focolaio di infezione respiratoria a Wuhan (Cina) è statosegnalato all’Organizzazione Mondiale della Sanità (OMS). L’infezione sostenuta dal patogeno SARS-CoV-2 identificato come causa di quella malattia (oggi descritta come COVID-19) si è velocemente diffusa a livello mondiale e, ad oggi, gli infettati hanno superato i 40 milioni con più di un milione di decessi.
Sono già disponibili molte soluzioni diagnostiche che identificano la presenza di SARS-CoV-2 nelle secrezioni respiratorie e che possono avere vari gradi di utilità per tracciare il virus e ad agire tempestivamente per sopprimere la trasmissibilità.
Nella crisi pandemica internazionale è essenziale che i saggi diagnostici disponibili siano però molto affidabili e accurati e ciò è cruciale per fornire assistenza medica tempestiva all’individuo infetto, così come a prevenire la diffusione del patogeno ad altri individui.
Infatti, un risultato dei test diagnostici falso negativo può portare alla diffusione dell’epidemia nella comunità; allo stesso modo il risultato falso positivo può portare a trattamenti non necessari e può incidere pesantemente sulla qualità di vita dei soggetti e di tutti coloro con cui hanno avuto contatto recentemente.
E’ pertanto fondamentale che l’urgenza e la necessità di disporre di test diagnostici rapidi non ne compromettano la qualità.
OBIETTIVO
Obiettivo di questo progetto è stato quello di svolgere un’analisi comparativa tra l’identificazione di SARS-CoV-2 eseguita con metodi molecolari standard (saggi con durata media di 5 ore e con un importante impegno di risorse umane) e quella ottenuta con la metodica miniaturizzata On-Site Real Time PCR Rapid
Shineway SWM-01, molto semplice da eseguire, utilizzabile anche come sistema POCT (Point Of CareTesting) e che permette di ottenere i risultati in circa un’ora.
DISEGNO SPERIMENTALE
Per l’analisi comparativa si sono utilizzati 145 campioni naso-faringei prelevati da pazienti mediante tamponi FLOQSwabs™ (COPAN) conservati nel terreno di trasporto universale UTM™ (COPAN) conservato presso la sezione Microbiologia e Virologia dell’ASST Spedali Civili di Brescia per svolgere le indagini diagnostiche routinarie. Dopo le analisi routinarie e la refertazione, i campioni appartenenti ai gruppi
“positivi” e “negativi” sono conservati subito utilizzati per l’analisi comparativa o, in alternativa, conservati in freezer a -80°C, senza essere sottoposti a ulteriori cicli di gelo-scongelo.
Nel momento in cui sono state eseguite le prove per la valutazione dello strumento Shineway gli acidi nucleici sono stati estratti utilizzando l’estrattore automatico “GenePure Pro–NPA32” (BIOER Technology) utilizzando i reattivi di estrazione “MagaBio plus Virus DNA/RNA purification kit II – BSC71S1E” (BIOER Technology), seguendo il protocollo del produttore.
L’amplificazione degli estratti è stata eseguita utilizzando il kit commerciale “SARS-CoV-2 Nucleic Acid Detec+on Kit (Fluorescence RT-PCR) – BSJ16M1” (BIOER Technology).
L’amplificazione Real Time PCR è stata eseguita con lo strumento: On-Site Real Time PCR – SWM-01.
In parallelo gli stessi campioni estratti sono stati rivalutati con altri test commerciali secondo protocollo standard su pia/aforma CFX Biorad.
RISULTATI e CONCLUSIONI
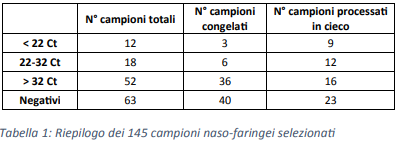
I 145 campioni sono stati selezionati all’interno di differenti coorti in base ai risultati restituiti dall’attuale metodica di laboratorio validata per la rilevazione di specifiche regioni del genoma di SARS-CoV-2 (Tabella 1).
Allo scopo di valutare la sensibilità dello strumento, la scelta è volutamente ricaduta su un alto numero di campioni con bassa carica virale.
In particolare, sono stati processati 45 campioni risultati positivi e 40 campioni negativi conservati in freezer a -80°C, senza essere sottoposti a cicli di gelo-scongelo.
A fresco, in contemporanea con le analisi routinarie, sono stati inoltre analizzati altri 60 campioni “in cieco”.
I risultati ottenuti con SWM-01 mostravano il 72% in concordanza con quelli rilevati dalle metodiche in uso.
La specificità, per tutti i campioni valutati , è risultata essere del 100%.
Analizzando i campioni abbiamo osservato un valore di sensibilità sempre superiore al 90% per i campioni rilevati sotto il Ct 32.
Il sistema si è quindi rivelato efficace nel rilevare la positività per tutti quei pazienti che, sintomatici e non, mostrano cariche virali compatibili con la trasmissibilità di SARS-CoV-2.
Inoltre, quando confrontati campioni valutati a fresco e campioni ottenuti dopo scongelamento non si è osservato nessun cambiamento significativo in termini di sensibilità (Tabella 2)
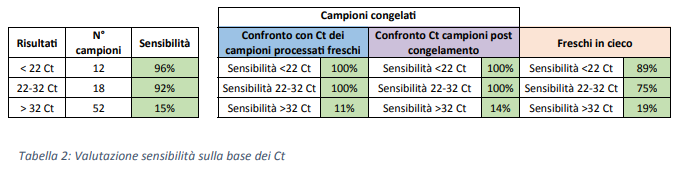
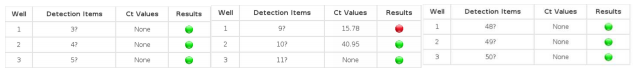
Figura 1: Esempio di schermata di risultati di campioni analizzati con sistema SWM-01
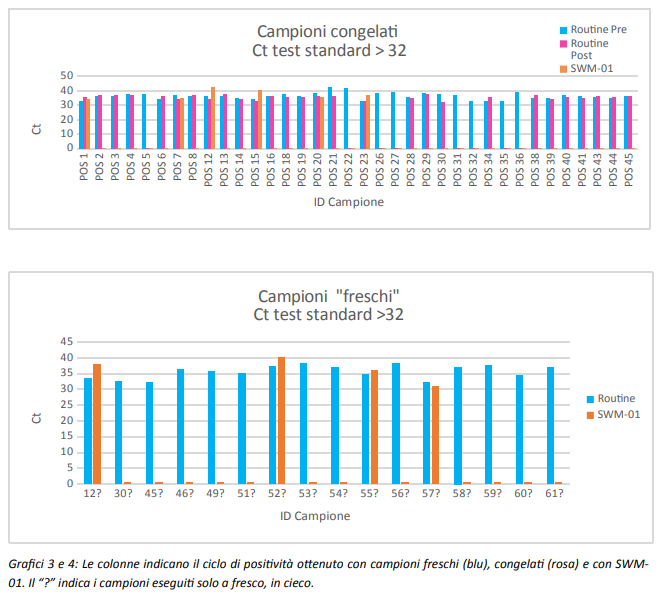
I grafici sotto mostrano, in de/aglio, il valore di Ct ottenuto per ogni singolo campione sottoposto all’analisi.
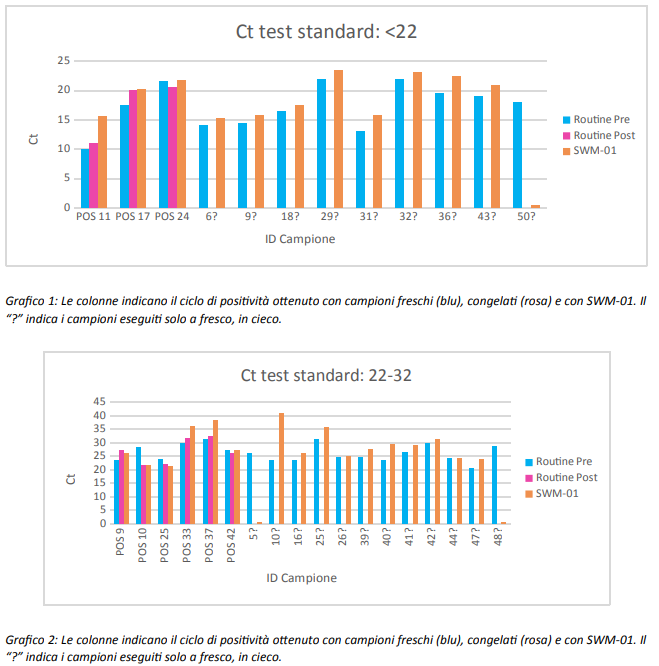
Dall’analisi “a posteriori” dei risultati ottenuti abbiamo identificato il Ct=32 come il ciclo soglia dei dati ottenuti dai test routinari atti a definire il risultato negativo ottenuto con SWM-01 affidabile.
Infatti solo 6/36 campioni congelati (17%) e 4/16 (25%) dei campioni processati a fresco, per i quali si è ottenuto un risultato con Ct>32 con le metodiche standard in uso, sono risultati positivi con SWM-01.
Il sistema analitico SWG-01 si è rivelato un ottimo test “point of care” con sensibilità sufficiente a identificare i portatori di SARS-CoV-2 potenzialmente infettanti con elevata specificità e buona sensibilità.
La presenza di un singolo target di PCR può far sfuggire campioni con alta carica virale che presentano però mutazioni (un campione discordante tra quelli ad alta carica virale ottenuti dall’analisi di routine).
L’implementazione con la rivelazione di target genici multipli può ovviare a questa criticità e, potenzialmente, innalzare la sensibilità generica del sistema.
Prof. Arnaldo Caruso – Presidente Società Italiana di Virologia


